En Çok Okunanlar
Özet
Çok ilaca dirençli (ÇİD) ve yaygın ilaç dirençli (YİD) Pseudomonas aeruginosa yüksek riskli klonlarının dünyada yayılımı küresel bir tehdittir. P. aeruginosa, kromozomal mutasyonların seçilmesi sonucu mevcut olan tüm antipsödomonal ilaçlara belirgin şekilde antimikrobiyal direnç geliştirme kapasitesine sahiptir. Tüm genom dizileme ile yapılan mutasyonel rezistom analizi, geniş çapta yayılım gösteren yüksek riskli klonların karakterizasyonuna ve antibiyotik direnç mekanizmalarının evrimsel dinamiklerinin anlaşılmasına imkân sağlamıştır. P. aeruginosa rezistom analizi, P. aeruginosa infeksiyonlarında tedavi stratejilerinin geliştirilmesine ve kullanılan antibiyotik tedavisinin etkinliğinin takibine olanak sağlayacaktır. Bu derlemede, P. aeruginosa rezistomunu biçimlendiren başlıca gen ve mutasyonlar ve P. aeruginosa’nın epidemik yüksek riskli klonları gözden geçirilmiştir.
Öne Çıkanlar
- Çok ilaca dirençli (ÇİD) ve yaygın ilaç dirençli (YİD) P. aeruginosa izolatları ile gelişen infeksiyonların prevalansında artışın nedenleri arasında; P. aeruginosa’nın özellikle hastanelerde ve bağışıklığı zayıflamış hastalarda ciddi infeksiyonlara yol açması, patojenin antimikrobiyal direnci yayma konusunda olağanüstü kapasiteye sahip olması ve P. aeruginosa’nın yüksek riskli klonlarının dünya çapında hızlı yayılım göstermesi gelmektedir.
- P. aeruginosa rezistomu, intrensek, mutasyonel ve horizontal kazanılmış rezistom olarak antibiyotik direnç genlerinin toplamını içermektedir.
P. aeruginosa rezistomundaki mutasyonel değişiklikler antibiyotiğe maruz kalma ile doğrudan ilişkilidir. Bir antibiyotiğe maruziyet o antibiyotik için P. aeruginosa rezistomunun olumsuz evrilmesine neden olurken, ortaya çıkan bu değişiklik bir diğer antibiyotiğin duyarlılığını olumlu yönde etkileyebilmektedir.
- ÇİD/YİD P. aeruginosa infeksiyonlarında, P. aeruginosa rezistomunun detaylı analizi yapılarak her hastada doğru tedavi stratejisinin geliştirilmesi ve ardından başlanan antibiyotik tedavisinin etkinliğinin takip edilmesi ile yepyeni ve çok kullanışlı bir tedavi yaklaşımının ortaya çıkışı mümkün olacaktır.
PSEUDOMONAS AERUGIONOSA’NIN TARİHÇESİ VE GENEL ÖZELLİKLERİ
Günümüzde Pseudomonas aeruginosa olarak adlandırılan bu fırsatçı bakteriyel patojen, kültürü sırasında besiyerinde oluşturduğu karakteristik mavi-yeşil renk nedeniyle tarih boyunca farklı isimler almıştır.
Sédillot, 1850 yılında cerrahi örtülerde oluşan renk değişikliğinin bir etkenle ilişkili olduğunu gözlemleyen ilk araştırmacı olmuştur. Fordos, 1860 yılında bu mavi renk değişikliğinin bir pigmentten kaynaklandığını ilk ortaya koyan araştırmacıdır. 1862 yılında Lucke, bu pigmenti çubuk şeklindeki mikroorganizmalarla ilişkilendiren ilk kişi olmuştur. P. aeruginosa, 1882 yılında Gessard’ın “Bandajların Mavi ve Yeşil Renkleri Üzerine” başlıklı yayını ile iki hastanın mavimsi yeşil irinli kutanöz yaralarından organizmanın saf kültür şeklinde ürediğini bildirmesine kadar izole edilememiştir. 1889 ve 1894 yılları arasında yayımlanan birkaç makalede, P. aeruginosa (Bacillus pyocyaneus) hastaların yaralarında mavi-yeşil pürülan akıntının nedeni olarak tanımlanmıştır. Akut veya kronik infeksiyona yol açan P. aeruginosa’nın tanımlanması ve yayılma yolları hakkında daha kapsamlı bilgilerin verildiği çalışma ise Freeman tarafından 1916 tarihinde yayımlanmıştır (1).
P. aeruginosa; nemli ortamlarda yaşayabilen, bir ucunda tek veya nadiren 2-3 adet kamçısı ile çok hareketli olan, tek, ikili veya bazen kısa zincirler halinde bulunan Gram-negatif bir basildir. Besin gereksinimi çok az olup standart buyyon, %5 koyun kanı içeren triptik soy agar, Columbia agar, çikolata agar ve MacConkey agar gibi katı besiyerlerinde kolayca üreyebilir. Katı besiyerinde koloni kenarlarından lokalize yayılma eğilimi gösterir, yeşil ve floresan pigment üretimi ve meyve kokusu gibi karakteristik özelliklere sahiptir. İlk izolasyon sonrasında, solunum sekresyonlarında sıklıkla kolonize olarak yerleşebilmektedir. Farklı çevresel ortamlarda bulunabilen bir organizmadır. Bitkiler, hayvanlar ve insanlar dahil olmak üzere çeşitli canlı kaynaklardan izole edilebilir. Minimum beslenme gereksinimi ile hayatta kalma ve çeşitli fiziksel koşulları tolere etme yeteneği, P. aeruginosa’nın hem toplumsal alanlarda hem de hastane ortamlarında uzun süreli yaşamasına olanak sağlamaktadır. Solunum yoluna ait tedavi ekipmanları, antiseptikler, sabun, lavabo, paspas, ilaç ve fizyoterapi-hidroterapi havuzları dahil olmak üzere hastane ortamında bulunan çeşitli kaynaklardan izole edilebilmektedir. Hastane dışı ortamlarda ise; yüzme havuzlarında, sıcak su banyo küvetlerinde, kontakt lens çözeltisinde ve toprakta kolaylıkla saptanabilmektedir. Doğada geniş dağılımı ve toplum kaynaklı infeksiyon oluşturma potansiyeli olmasına rağmen, P. aeruginosa ile ilişkili ciddi infeksiyonlar genellikle hastane kaynaklıdır (2).
Son zamanlarda, çok ilaca dirençli (ÇİD) ve yaygın ilaç dirençli (YİD) P. aeruginosa izolatlarının ortaya çıkmasının ve yayılmasının küresel halk sağlığı açısından tehdit oluşturarak endişe verici hale gelmesinin birkaç ana nedeni bulunmaktadır. Öncelikle, P. aeruginosa, özellikle hastanelerde bulunan bağışıklığı zayıflamış hastalarda ciddi infeksiyonlara neden olmaktadır. Diğer yanda antimikrobiyal direnci yayma konusunda olağanüstü bir kapasiteye sahiptirler ve P. aeruginosa’nın yüksek riskli klonları dünya çapında hızlı bir şekilde yayılmaktadır (3).
P. aeruginosa izolatları arasında kombine direncin sık olduğu bilinmektedir. Avrupa Hastalık Önleme ve Kontrol Merkezi (“European Centre for Disease Prevention and Control – ECDC”) tarafından 2015 yılında P. aeruginosa izolatlarının %13.7’sinin en az üç ve %5.5’inin ise en az beş antimikrobiyal ilaç grubuna karşı dirençli olduğunu bildirilmiştir. Terapötik alternatiflerin olmaması, antibiyotiklere dirençli bu bakterilerin neden olduğu infeksiyonların morbidite ve mortalite açısından dünya çapında önemli bir tehdit oluşturduğu anlamına gelmektedir. ECDC’nin 2015 yılı verilerine göre, P. aeruginosa; pnömonilerin %30’undan, idrar yolu infeksiyonlarının %19’undan ve kan dolaşımı infeksiyonlarının %10’undan sorumludur (4). Dünya Sağlık Örgütü (DSÖ) 2017 yılında yayımlanan raporunda bu patojen için yeni antibiyotiklere acil olarak ihtiyaç duyulduğunu bildirmiştir (5).
PSEUDOMONAS AERUGINOSA REZİSTOMU
P. aeruginosa izolatları; çoklu kromozomal determinantlarının yanı sıra intrensek ve adaptif dirençle ilgili karmaşık düzenleyici yollar da dahil olmak üzere dikkate değer birçok antibiyotik direnç mekanizmasına sahiptir. Bu mekanizmalar aşağıda başlıklar halinde verilmiştir.
İntrensek (Doğal) Rezistom
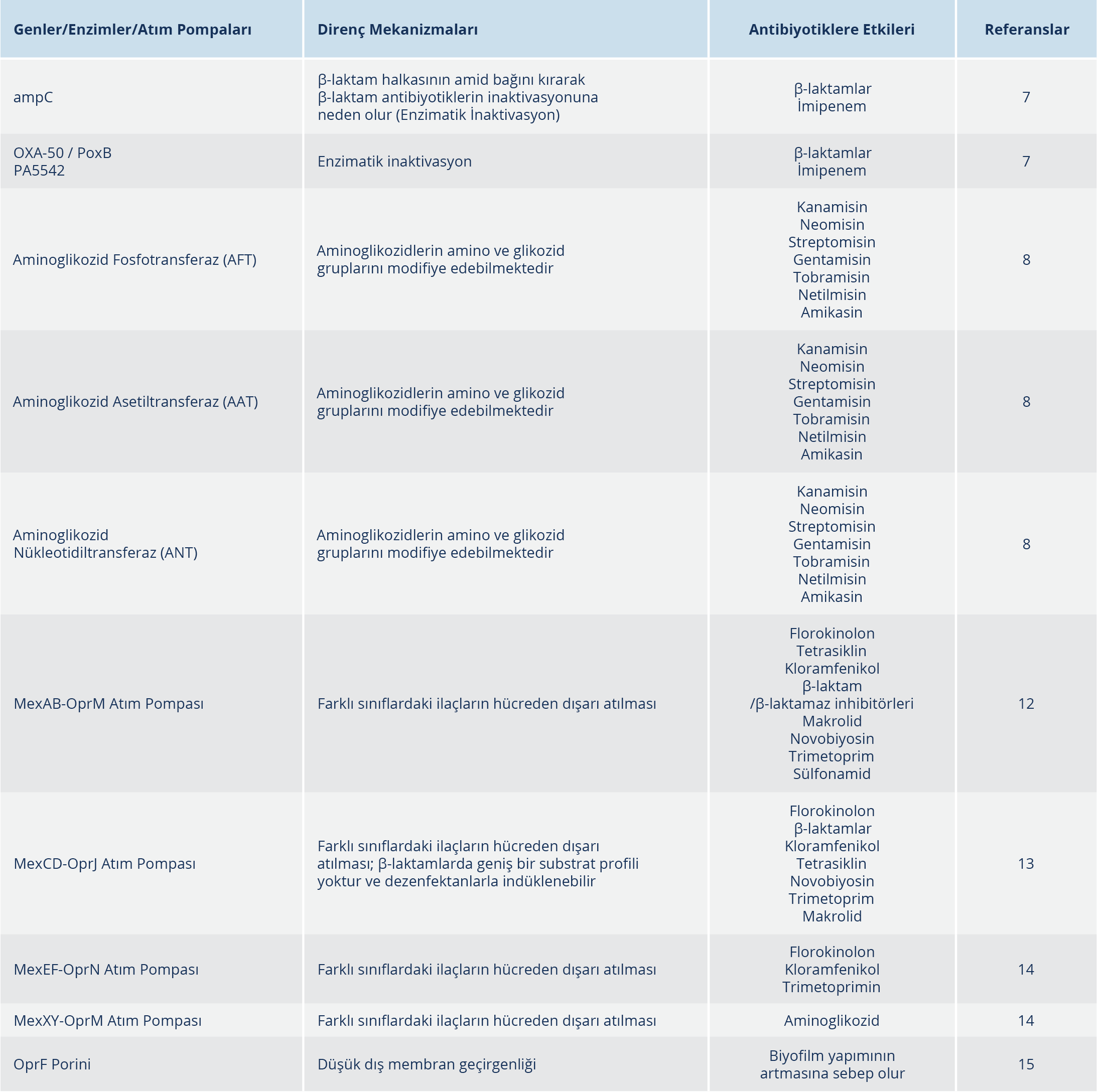
P. aeruginosa’nın diğer Gram-negatif mikroorganizmalara kıyasla doğal rezistom üzerinde büyük etkiye sahip olduğu düşünülen mekanizmaları bulunmaktadır. Bu mekanizmalar; indüklenebilir AmpC sefalosporinaz ekspresyonu gibi antibiyotikleri inaktive eden enzimlerin sentezlenmesi, temel (MexAB-OprM) ve indüklenebilir (MexXY) dışa atım pompası üretimi ve düşük dış membran geçirgenliğidir (Tablo 1) (6).
Antibiyotikleri İnaktive Eden Enzimler: Antibiyotikleri parçalayan veya yapılarını bozan antibiyotik inaktive edici enzimlerin üretimi, bakterilerdeki en önemli doğal direnç mekanizmalarından biridir. β-laktam ve aminoglikozid gibi antibiyotikleri modifiye eden birçok enzim P. aeruginosa tarafından yaygın olarak üretilmektedir. Antibiyotikleri inaktive eden enzimler, hidroliz olabilen amidler ve esterler gibi kimyasal bağlara etki eder (6).
Diğer Gram-negatif bakteriler gibi P. aeruginosa da hidrolitik β-laktamaz enzimini kodlayan indüklenebilir ampC genine sahiptir. Bu enzim β-laktam halkasının amid bağını kırarak β-laktam antibiyotiklerin inaktivasyonuna yol açmaktadır. İndüklenebilir AmpC ekspresyonu, P. aeruginosa’nın imipeneme karşı doğal azalmış duyarlılığında belirleyici bir rol oynamaktadır. Çünkü bu antibiyotiğin hidrolitik stabilitesi, yüksek indükleyici potensinden etkilenmektedir. Diğer iki kromozomal β-laktamaz, OXA enzimi OXA-50 / PoxB ve daha yakın zamanda açıklanan imipenemazın (PA5542) intrensek dirençte rol oynadığı bildirilmiş olmakla birlikte daha net çalışmaların yapılması gerektiği araştırmacılar tarafından vurgulanmaktadır. Sonuç olarak, indüklenebilir β-laktamaz üretimi, P. aeruginosa’nın bu ajanlara karşı doğal direncinde anahtar bir role sahiptir (7).
Antibiyotik direncinin 1960’lara kadar ortaya çıkmasına ve ökaryotik hücrelere karşı yüksek toksisiteye sahip olmasına rağmen; aminoglikozidler (amikasin, gentamisin, kanamisin, neomisin, netilmisin, paromomisin, streptomisin ve tobramisin) uzun yıllardır P. aeruginosa infeksiyonlarının tedavisinde kullanılmaktadır. Günümüzde, P. aeruginosa’da aminoglikozidlere direnç çok yaygın olup dünyanın her yerinden bildirilmektedir. Aminoglikozid grubu antimikrobiyaller, glikozid bağlarla amino şekerlere bağlanan aminosiklitol halkasına sahip antimikrobiyal ilaç gruplarıdır. İnaktive edici enzimler, aminoglikozidlerin aktivitesi için hayati önem taşıyan molekülün spesifik amino ve glikozid gruplarını modifiye edebilmektedir. P. aeruginosa’da reaksiyon türlerine ve etkiledikleri fonksiyonel gruplara göre, üç aminoglikozid modifiye edici enzim formu tanımlanmıştır. Bunlar; aminoglikozid fosfotransferaz (AFT), aminoglikozid asetiltransferaz (AAT) ve aminoglikozid nükleotidiltransferaz (ANT)’dır. Söz konusu enzimler; kanamisin, neomisin, streptomisin, gentamisin, tobramisin, netilmisin ve amikasin antibiyotiklerinde sıklıkla dirence neden olmaktadır (8).
Dışa Atım Pompaları: Dışa atım pompaları kaynaklı bakteriyel rezistom, antimikrobiyal ilaçları zardan sitoplazmaya girdikten sonra hücreden dışarı atan sistemlerdir. Bu sistemler, tek veya birden fazla antibiyotik sınıfını dışarı atabilmektedir. Atım pompaları, hem Gram-pozitif hem de Gram-negatif bakterilerde farklı şekillerde ifade edilebilir (9). Günümüzde dışa atım pompa sistemleri en iyi P. aeruginosa izolatlarında çalışılmış olup yapılarına (amino asit sekans homolojisi) ve enerji gereksinimlerine göre altı farklı üst aileye ayrılmıştır:
- Majör kolaylaştırıcı süper ailesi [“major facilitator superfamily (MFS)”],
- Küçük çoklu ilaç direnç protein ailesi [“small multidrug resistance (SMR) protein family”],
- Çoklu ilaç ve toksik bileşik parçalama ailesi [“multidrug and toxic compound extrusion (MATE) family”],
- Direnç nodülasyon bölümü süper ailesi [“resistance-nodulation-division (RND) superfamily”],
- Adenozin 3’-trifosfat (ATP) bağlayan kaset süper ailesi [“ATP-binding cassette (ABC) superfamily”],
- Proteobakteriyel antimikrobiyal bileşik dışa atım süper ailesi [“proteobacterial antimicrobial compound efflux (PACE) superfamily”] (10).
P. aeruginosa izolatlarında yapılmış olan genom dizi analizlerinde, yukarıdaki 6 aile için de atım sistemleri gösterilmiş olmakla birlikte pompaların çoğu RND ailesine ait olup 12 farklı RND sistemi tanımlanmış durumdadır. Bu protein yapıda olan RND dışa atım pompaları, sitoplazmik membran taşıyıcıları, periplazmik bağlayıcı proteinler ve dış membran porin proteinlerinden oluşmakta olup günümüzde patojenin büyük bir sağlık sorunu haline gelen antibiyotik direncinde önemli rol oynamaktadır (11). P. aeruginosa rezistomunda yer alan başlıca dört RND süper ailesi bulunmaktadır. Bunlardan ilk keşfedilen MexAB-OprM atım pompası olup florokinolon, tetrasiklin, kloramfenikol, β-laktam/β-laktamaz inhibitörlerinin yanı sıra makrolid, novobiyosin, trimetoprim ve sülfonamidler gibi farklı sınıflardaki ilaçların hücreden dışarı atılmasını sağlamaktadır (12).
Bir diğer atım pompası; florokinolon, β-laktam, kloramfenikol, tetrasiklin, novobiyosin, trimetoprim ve makrolidler gibi çeşitli antimikrobiyal ilaçların hücreden dışarı atılmasını sağlayan MexCD-OprJ’dir. Bu sistemde, ilk keşfedilen MexAB-OprM’den farklı olarak β-laktamlarda geniş bir substrat profili yoktur. Ayrıca, MexCD-OprJ’nin dezenfektanlarla indüklenmesi önemli bir faktördür. Bu sonuçlar, dezenfaktanların sık olarak kullanıldığı hastanelerde MexCD-OprJ’nin P.aeruginosa’daki intrensek çoklu ilaç direncinde önemli rol oynadığını göstermektedir (13). 1997 yılında tanımlanan MexEF-OprN pompası; florokinolonlar, kloramfenikol ve trimetoprimin hücreden dışarı atılmasını sağlamakla birlikte pompanın, kullanımda olan β-laktamlara belli bir afinitesi yoktur. Sonuncu RND süper ailesine ait dışa atım pompası ise aminoglikozid direncine yol açan MexXY-OprM’dir (14).
Düşük Dış Membran Geçirgenliği: P. aeruginosa’nın dış membran geçirgenliği son derece kısıtlıdır ve Escherichia coli’ye kıyasla %60 oranında daha düşüktür. OprF porini, P. aeruginosa’nın baskın porini olup kapalı bir sistemdir. İzolatlarda kapalı OprF kanallarının varlığı, P. aeruginosa’nın dış membran geçirgenliğinin neden diğer bakterilerden daha düşük olduğunu açıklamaktadır. Ayrıca P. aeruginosa’da, OprF’nin varlığının biyofilm yapımının artmasına sebep olduğu düşünülmektedir (15).
Mutasyonel Rezistom
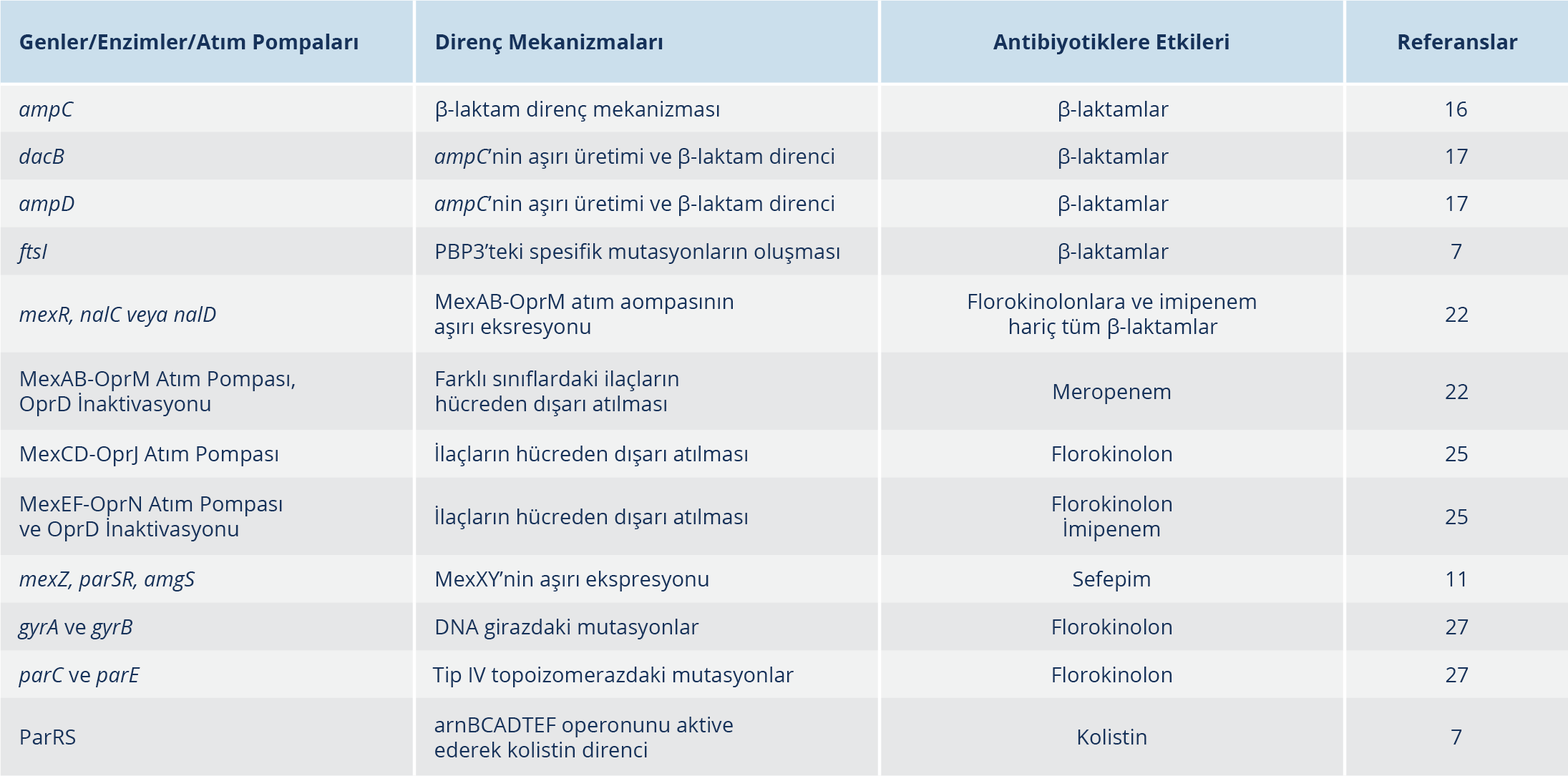
P. aeruginosa, geniş intrensek rezistomunun yanı sıra, kromozomal mutasyonların kazanılması yoluyla mevcut tüm antibiyotiklere karşı daha fazla antimikrobiyal direnç geliştirme konusunda olağanüstü bir yetenek sergilemektedir (Tablo 2). P. aeruginosa izolatlarında; direnç düzeylerini artırdığı ve böylece mutasyonel rezistoma şeklini verdiği bilinen çok sayıda gen ve mutasyon tanımlanmış durumdadır. Geniş bir gen yelpazesini içeren kromozomal ampC sefalosporinazın aşırı üretimi, P. aeruginosa klinik izolatlarının %20’sinden fazlasında saptanmış durumda olup mutasyon kaynaklı en yaygın β-laktam direnç mekanizmasını oluşturmaktadır. Penisilin bağlayan protein (PBP) 4’ü kodlayan dacB’nin ve N-asetilmuramil-L-alanin amidazı kodlayan ampD’nin mutasyonel inaktivasyonlarının, ampC’nin aşırı üretimi ve β-laktam direncinin en yaygın mekanizmaları olduğu bilinmektedir. Ayrıca, PBP4 inaktivasyonunun, CreBC / BlrAB iki bileşenli sistemi aktive ettiği ve direnç seviyelerini daha da artırdığı gösterilmiştir (16, 17). Son zamanlarda transkripsiyonel regülatör AmpR’nin yapısının modifikasyonuna yol açan spesifik mutasyonlar klinik P. aeruginosa izolatlarında tespit edilmeye başlamış olup β-laktam direncine neden olan bir diğer mutasyonel rezistom basamağını oluşturmaktadır. Bu mutasyonlar, daha önce yapılmış olan çalışmalarda P. aeruginosa dışındaki mikroorganizmalarda tespit edilmiş olan D135N ve özellikle kistik fibrozis (KF) hastalarında bildirilmiş olan epidemik ÇİD ST175 yüksek riskli klonla ilişkili R154H mutasyonlarıdır. Yapılan çalışmalar AmpC aşırı üretiminin yanı sıra, AmpC’nin yapısal modifikasyonuna yol açan mutasyonların, yeni β-laktam/β-laktamaz inhibitör kombinasyonları olan seftolozan/tazobaktam ve seftazidim/avibaktam dahil olmak üzere β-laktamlara dirence neden olabileceğinin altını çizmiştir (18).
Benzer şekilde başka bir çalışmada da P. aeruginosa klinik izolatlarının küçük bir bölümünde (yaklaşık %1) seftolozan/tazobaktam ve seftazidim/avibaktam direncine yol açan, genişlemiş spektrumlu AmpC yapımına neden olan birkaç amino asit değişikliğine sahip varyantlar tespit edilmiştir. Genişlemiş spektrumlu AmpC’ler, hipermutasyona neden olmaksızın özellikle KF ve kronik obstrüktif akciğer hastalarında sık görülmektedir (19).
Bugüne kadar seftolozan/tazobaktam ve seftazidim/avibaktam direncinde artışa neden olan 300’ün üzerinde Pseudomonas türevi sefalosporinaz (PTS) varyantı bildirilmiştir. PTS varyantlarının güncellenmiş veri tabanına, Antonio Oliver’ın liderliğindeki Bakteriyel İnfeksiyonların Antibiyotik Direnci ve Patojenitesi Grubu’nun internet sayfasından ücretsiz olarak ulaşılabilmektedir (20). β-laktamazlara ek olarak, PBP modifikasyonu da β-laktam direncindeki artışa neden olmaktadır. Özellikle ftsI tarafından kodlanan PBP3’teki mutasyonlar sıklıkla bu izolatlarda artışa neden olmaya başlamıştır. KF hastaları, epidemik suşlar ve in vitro çalışmalardan elde edilen son veriler, PBP3’teki spesifik mutasyonların β-laktam direncinin ortaya çıkmasında rol oynadığını göstermiştir. Bu dirence neden olduğu bildirilen en sık mutasyonlar, β-laktam PBP3 inaktivasyon kompleksinin stabilizasyonunda yer alan alanlarda bulunan R504C / R504H ve F533L’dir (7). Karbapeneme özgü porin olan OprD’nin kaybı, ya oprD genindeki inaktive edici mutasyonların ya da atım pompa sistemi olan MexEFOprN veya CzcCBA’nın oprD ekspresyonunu artıran mutasyonların bir sonucu olabilir. OprD porinin mutasyonel inaktivasyonu veya aşağı regülasyonu özellikle indüklenebilir AmpC üretimi ile birlikte gerçekleştiğinde, imipenem direncinde ve azalmış meropenem duyarlılığında ciddi bir artış görülmektedir. İmipenem direncinin prevalansı dünya çapında %20’nin üzerindedir ve bu izolatların çoğunda OprD porininin eksik olduğu bildirilmektedir (7, 21, 22).
OprD inaktivasyonu, tüm klasik anti-psödomonal β-laktamlara karşı direnci artırmak için sıklıkla AmpC aşırı ekspresyonu ile sinerjik olarak hareket etmektedir (23). P. aeruginosa’nın dört ana atım pompasından birinin mutasyon kaynaklı aşırı ekspresyonu da mutasyonel rezistomda önemli bir rol oynamaktadır. MexAB-OprM (%10) ve MexXY’nin (%30) aşırı ekspresyonu klinik izolatlar arasında yaygınken MexCD-OprJ ve MexEF-OprN aşırı ekspresyonunun prevalansı (%5) önemli ölçüde daha düşüktür (21).
MexAB-OprM, en geniş substrat spektrumuna sahip olup bu dışa atım pompasının mutasyon kaynaklı aşırı ekspresyonu, florokinolonlara ve imipenem hariç tüm β-laktamlara olan duyarlılığın azalmasına neden olmaktadır. MexAB-OprM atım pompasının aşırı ekspresyonu ve OprD inaktivasyonunun birlikte olması, klinik suşlar arasında meropeneme direncin başlıca nedenlerinden biridir (22). MexXY’nin intrensek aminoglikozid direncindeki rolünün yanı sıra, mutasyonuna bağlı aşırı üretimi de klinik izolatlarda sefepime direncin yaygın bir nedeni olarak ortaya çıkmaktadır (24). MexCD-OprJ veya MexEF-OprN aşırı üretiminin yaygınlığı daha düşük olup esas olarak florokinolonlara direnci etkilemektedir. Ancak MexEF-OprN’nin aşırı üretimini tetikleyen mutasyonlar (mexT/mexS) oprD’nin baskılanması nedeniyle imipeneme direnci de belirlemektedir (25). MexAB-OprM’nin aşırı ekspresyonuna yol açan, regülasyonunda görevli olan mexR, nalC veya nalD genlerindeki mutasyonlar imipenem dışında kalan birçok β-laktam antibiyotiğe karşı dirence neden olurken, mexZ, parSR, amgS genlerindeki mutasyonlar sonucu ortaya çıkan MexXY’nin aşırı ekspresyonu özellikle sefepim direncine neden olmaktadır (11). Özellikle kronik infeksiyonlarda yaygın olan MexCD-OprJ’nin aşırı ekspresyonu, muhtemelen diğer direnç mekanizmaları ile antagonistik etkileşime bağlı olarak β-laktam ve aminoglikozidlere artan duyarlılığa neden olmasına rağmen, sefepim minimal inhibitör konsantrasyon (MİK) değerlerinin artmasına da neden olmaktadır. MexCD-OprJ’nin aşırı ekspresyonu, majör yapısal (MexAB-OprM) ve indüklenebilir (MexXY-OprM) atım pompaları ile ortaya çıkabilir. AmpC β-laktamaz yapımına neden olarak P. aeruginosa’da intrensek direncin bozulmasına yol açmaktadır (26). Dışa atım pompalarının aşırı ekspresyonunun yanı sıra, P. aeruginosa izolatlarında florokinolon direnci sıklıkla DNA giraz (gyrA ve gyrB) ve tip IV topoizomeraz (parC ve parE) enzimlerindeki mutasyonlardan kaynaklanmaktadır (27). Florokinolon direncinin prevalansı coğrafik bölgeye göre değişmekle birlikte, birçok ülkede %30 ila %40’ın üzerindedir. Yüksek düzey florokinolon direnci P. aeruginosa’nın yüksek riskli klonlarında yaygındır ve genellikle GyrA-T83 ve ParC-S87 mutasyonlarının birlikte olması ile ortaya çıkar (28). Klinik izolatlar arasında MexAB-OprM ve MexXY-OprM’nin aşırı üretimi daha sıktır (27). Son zamanlarda yapılan çalışmalar, özellikle KF hastalarında MexXY atım pompasının aşırı üretimine ve yatay olarak edinilen mekanizmalara ek olarak, aminoglikozid direncinin fusA1’deki mutasyonlarından kaynaklanabileceğini göstermektedir (29). Son olarak, P. aeruginosa’da kolistin direncinin prevalansı nispeten diğer antimikrobiyal ilaçlara göre daha düşük (%5) olsa bile, son zamanlarda ÇİD/YİD izolatlarının neden olduğu infeksiyonların tedavisinde son çare olarak kullanılan kolistinin artan kullanımı nedeniyle bu ilaca karşı direnç artmış durumdadır. Kolistin direnci sıklıkla, 4-amino-4-deoksi-L-arabinoz ilavesini takiben lipopolisakkaridin (LPS) lipit A kısmının modifikasyonundan kaynaklanmaktadır. Bu mutasyonlar sıklıkla, arnBCADTEF operonunun aktivasyonuna yol açan PmrAB ve PhoPQ ile bağlantılıdır. Yakın zamanda yapılmış olan bir çalışmada; ParRS iki bileşenli regülatördeki mutasyonların, sadece arnBCADTEF operonunu aktive ederek kolistin direncini tetiklemekle kalmayıp, MexXY’nin aşırı üretimine ve OprD’nin aşağı regülasyonu yoluyla bir ÇİD profiline de yol açtığı gösterilmiştir (7). Yüksek düzey kolistin direncinde mutS yokluğu gösteren mutatör fenotiplerin ortaya çıkışı da rol oynamaktadır (30).
Horizontal Rezistom
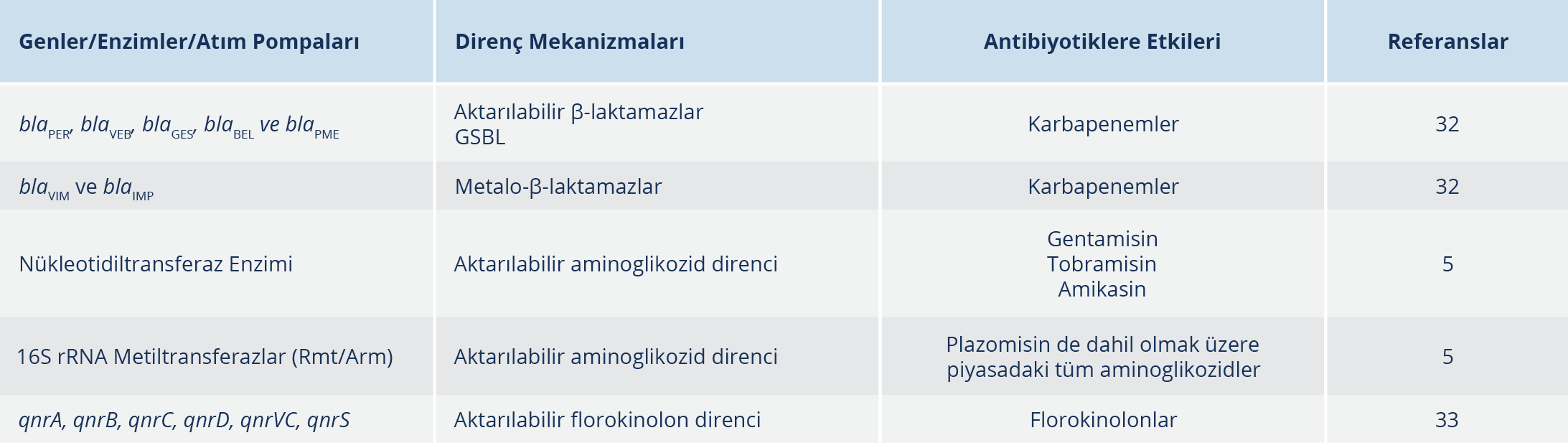
Nispeten en sık görülen mutasyonel rezistoma ek olarak, P. aeruginosa’da horizontal rezistomda hızlı bir artış görülmektedir. Aktarılabilir β-laktamazlar, genişlemiş spektrumlu β-laktamaz (GSBL)’lar ve karbapenemazlar dünya çapında hızlı bir şekilde artarak yayılmaktadır. Bu dağılım hastane ve coğrafik bölgeye bağlı olarak %1-50 arasında değişmektedir. Ne yazık ki, P. aeruginosa’da aktarılabilir β-laktamazları tespit etme zorluğundan dolayı prevalans oranları birçok bölgede eksik değerlendirilmektedir. GSBL’leri ve karbapenemazları kodlayan genler genellikle sınıf 1 integronlarda aminoglikozid direncinin belirleyicileriyle birlikte bulunur. Bu integronlar, genellikle bakteriyel kromozom üzerinde yer alan transpoze edilebilir elemanlara eklenir (31, 32).
P. aeruginosa izolatlarında en sık bildirilen GSBL’ler, sınıf D (OXA-2 veya OXA-10 varyantları) ve sınıf A (blaPER, blaVEB, blaGES, blaBEL ve blaPME) genleridir. Tipik olarak enterik bakterilerde saptanan TEM, SHV veya CTX-M β-laktamazlar gibi sınıf A GSBL’ler, P. aeruginosa’da nadiren görülmektedir. P. aeruginosa’da blaVIM ve blaIMP genleri başta olmak üzere metalo-β-laktamazlar (MBL) en yaygın bildirilen karbapenemazlardır. Farklı coğrafi bölgelerde blaGIM, blaNDM ve blaFIM genleri nadiren tespit edilebilmektedir (32).
Aktarılabilir aminoglikozid direnci en sık olarak, sınıf 1 integronlarda kodlanan aminoglikozid modifiye eden enzimler tarafından gerçekleşmektedir. P. aeruginosa izolatlarında en yaygın olarak tespit edilen enzimler, gentamisin, tobramisin ve amikasin direncine yol açan nükleotidiltransferaz enzimidir. Bununla birlikte, yeni bir aminoglikozid olan plazomisin de dahil olmak üzere piyasadaki tüm aminoglikozidlere direnç kazandıran 16S rRNA metiltransferazlar (Rmt/Arm) aminoglikozid direncinde rol oynayan önemli tehditlerden biridir (Tablo 3) (5).
Qnr genleri ile plazmide bağlı florokinolon direnci ortaya çıkmaktadır. Tanımlanan farklı genler; qnrA, qnrB, qnrC, qnrD, qnrVC ve qnrS’dir. Qnr belirleyicileri (QnrVC1) tarafından oluşan aktarılabilir florokinolon direnci zaman zaman farklı coğrafik bölgelerde tespit edilmektedir. Çok yakın tarihli bir çalışmada, yeni bir fosfotransferaz (CrpP) tarafından ortaya çıkan plazmit aracılı kinolon direncinin olduğu bildirilmiştir (33).
Yeni seftolozan/tazobaktam ve seftazidim/avibaktam kombinasyonlarının, seftolozan/tazobaktamda seftolozanın AmpC tarafından hidrolizine karşı stabil olması ve seftazidim/avibaktamda avibaktamın AmpC’ye karşı inhibe edici aktivitesinin varlığı, bu ilaç kombinasyonlarını AmpC hidrolizine karşı nispeten stabil hale getirmektedir (19).
P. aeruginosa’da mutasyonel rezistomun analizi sonucu horizontal olarak kazanılmış direnç genlerinin antibiyotik direnç genotipini ortaya koyması, antibiyotik direnç fenotipinin belirlenmesi açısından önemlidir. Böylece, yeni tedavi stratejilerinin tasarlanması ve uygulanan antibiyotik tedavisinin etkinliğinin takip edilmesi mümkün olacaktır. Antibiyotik etkisi altında, o antibiyotik için mutasyonel rezistomun evrimleşmesi beklenen bir gelişme olmakla birlikte, böyle bir koşul altında farklı direnç mekanizmaları arasındaki etkileşimin direnç profilini nasıl etkilediğinin anlaşılması çok daha kompleks bir durumdur (29). Benzer şekilde çapraz direnç (kollateral direnç), bir antibiyotik etkisine maruz kalma durumunda aynı veya benzer sınıftaki bir diğer antibiyotiğe direnç gelişiminin de söz konusu olacağını işaret etmektedir. Bir antibiyotiğe maruziyet durumunda diğer antibiyotiğe duyarlılığın artışı olarak bilinen kollateral duyarlılıkta ise iki ayrı mekanizma rol oynamaktadır (34, 35). Bu mekanizmalardan biri, antibiyotiğe maruziyetin doğrudan başka bir antibiyotiğe duyarlılığı artırmasıdır. Örneğin; AmpC tipi β-laktamaz mutasyonları, sefalosporinlerin hidrolizini artırırken penisilin veya karbapenem hidrolizini azaltmaktadır (36). İkinci mekanizma ise, ortaya çıkan direnç mekanizmasının bir diğer direnç mekanizmasının aktivitesini bozmasıdır. Buna örnek olarak atım pompaları arasındaki yarışma verilebilir. Bir atım pompasının aşırı ekspresyonu bir diğerinin ekspresyonunda bozulmaya neden olabilir. (26). Bu nedenle, mutasyonel rezistomun evrimleşmesi yalnız tedavide kullanılan tek bir antibiyotiğe değil, beraberinde hatta daha önce kullanılmış olan antibiyotiklere bağlı olarak da şekillenmektedir (37). Bu durum, bakterinin türüne ve kullanılan antibiyotiğe göre değişiklik göstermektedir. Tedavide kullanılan bir antibiyotik gelecekte kullanılacak olan antibiyotikler için de direnç rezistomunun evrimleşmesine neden olmaktadır.
EPİDEMİK YÜKSEK RİSKLİ KLONLAR
P. aeruginosa’nın küresel yayılım gösteren yüksek riskli klonları ÇİD/YİD fenotipleri ile ilişkilidir. P. aeruginosa’da küresel yayılımlarına, ÇİD/YİD profilleriyle olan ilişkilerine ve ayrıca GSBL, karbapenemaz gibi horizontal kazanılan β-laktamazlara bağlı olarak dünyada var olan yüksek riskli 10 klon tanımlanmış durumdadır. Bu klonlar ST235, ST111, ST175, ST233, ST244, ST357, ST308, ST277, ST654 ve ST298’dir (38). Söz konusu klonların üçü (ST235, ST111, ST175) dünya çapında hızla artmakta olup son zamanlarda sıklıkla izole edilmektedir. Ancak bu yüksek riskli klonlara özgü mutasyonel ve kazanılmış direnç mekanizmaları hakkında hala çok az bilgi bulunmaktadır (39).
P. aeruginosa’da ST235 Yüksek Riskli Klonu
Üç yüksek riskli klondan O11 serotipi ile ilişkili olan ST235 en yaygın olan klondur ve beş kıtaya yayılmış bir şekilde birçok ülkede bulunmaktadır (40). ST235 klonu, hipervirülan fenotipe sahip olup 60 farklı β-laktamaz varyantına ve sınıf A /sınıf B karbapenemazlara sahiptir. ST235 klonunun, integronlar, transpozonlar ve plazmitler dahil olmak üzere horizontal rezistom determinantları ile ilişkisi çok büyüktür (3). ExoU sitotoksin üretimi, ST235 klonunun hastane infeksiyonlarında yüksek mortalite göstermesinden sorumludur. Farklı ülkelerden 27 yıllık bir dönemde toplanan örneklerin 2018 yılında yapılan genomik analizi sonucunda; homolog rekombinasyonda rol oynayan DprA determinantının ST235 klonunda spesifik olarak var olmasının, yüksek riskli bu klonu diğer P. aeruginosa klonlarından ayıran en önemli özellik olduğu belirlenmiştir. DprA, kazanılmış DNA’nın konak kromozomuna doğal olarak transforme olması için gerekli olan rekombinaz RecA’nın integrasyonunda gereklidir. DprA, dönüşebilen türlerde homolog rekombinasyondan sorumlu DNA işlemci proteini olup bu proteinin varlığı P. aeruginosa ST235 klonunda bakterinin yabancı direnç elemanlarını kazanmasına neden olarak yüksek riskli klonun devamını sağlamaktadır (41).
ST235 yüksek riskli klonlarında A, B ve D sınıflarından 39 farklı kazanılmış β-laktamaz saptanmış durumdadır. Bu klon, birçok A sınıfı GSBL içermektedir ancak aralarında PER-1 coğrafik olarak en yaygın olanıdır. Bununla birlikte, B sınıfı karbapenemazlar, IMP ve VIM varyantları dahil olmak üzere ST235 izolatları arasında çok daha sıktır. Ancak VIM-2 bu yüksek riskli klon arasında coğrafi olarak en yaygın olan β-laktamazdır. Sınıf D enzimler içinde özellikle OXA-2 ve OXA-17, ST235 klonunda sıklıkla bildirilmektedir (3). ST235 klonunda kanamisin, tobramisin ve amikasin direncine yol açan aac(6’)-Ib aminoglikozid asetiltransferaz bildirilmiş durumdadır. Avrupa’daki ST235 klonuna ait suşlar genellikle aminoglikozid 6’-N-asetiltransferazı kodlayan aacA7 içermekte ve bunların bazıları aph(6)-Id aminoglikozid O-fosfotransferaz veya aadA1 adenililtransferaz taşımaktadır (38, 42).
P. aeruginosa’da ST111 Yüksek Riskli Klonu
ST235 klonunu ikinci sırada takip eden ve serotip O12 ile ilişkili olan ST111 ise, sıklıkla beraberinde VIM-2 karbapenemaz yapımı gösterir ve dünya çapında bir dağılıma sahiptir; şimdiye kadar Okyanusya hariç her kıtadan bildirilmiştir (40). İspanya’da bulunan 10 farklı hastanenin katılımı ile gerçekleşen çok merkezli çalışmaya dahil edilen 190 izolatın %10.5’i YİD, %23’ü ÇİD ve %24’ü bir veya iki antimikrobiyal ilaca karşı dirençli (orta dirençli), %43’nün ise test edilen tüm antipsödomonal ilaçlara karşı duyarlı olduğu tespit edilmiştir. Çalışmadaki YİD izolatlarının birinde, ÇİD izolatlarının ikisinde ve orta dirençli izolatların birinde ST111 klonunun varlığı tanımlanmıştır. Çalışmadaki ST111 yüksek riskli klonuna ait dört izolatta kazanılmış β-laktamazların varlığı tespit edilmiştir. ST111 klonuna ait üç izolatta (YİD/ÇİD) VIM-2 ve OXA-46; orta dirençli olan ST111 klonuna ait bir izolatta ise dar spektrumlu penisilinaz olan PSE-1 pozitif bulunmuştur. İlginç bir şekilde, birbirine uzak iki farklı hastaneden temin edilmiş olan YİD/ÇİD ST111 klonuna ait üç izolatta aynı spesifik inaktive edici OprD (W339X) mutasyonunun yanı sıra yüksek düzey florokinolon direncine neden olan iki mutasyonun (GyrA T83I ve ParC S87L) varlığı da gösterilmiştir (18, 39). Columbia’da yayımlanmış olan bir raporda, ST111 klonuna ait bir P. aeruginosa suşunda blaVIM-2 ve blaKPC-2’nin birlikte bulunduğu ve amikasin, siprofloksasin, polimiksin ile karbapenem ve aztreonam dahil tüm β-laktamlara dirençli olduğu bildirilmiştir (42). 2015 yılında Birleşik Krallık’ta ST111 klonuna ait 87 P. aeruginosa izolatının analiz edildiği çalışmada ise; test edilen izolatların çoğunun (n=74) blaVIM-2, bazılarının ise blaIMP-1 ve blaIMP-13 pozitif, bir suşun ise blaNDM-1 pozitif olduğu tespit edilmiştir (43).
P. aeruginosa’da ST175 Yüksek Riskli Klonu
ST175 klonu, ilk olarak 2005 yılında Birleşik Krallık ve Kanada’da tanımlanmıştır (42). Serotip O4 ile ilişkili olan ST175, birçok Avrupa ülkesinde geniş çapta yayılım gösteren düşük virulansa sahip bir klondur. Ancak, AmpC’nin aşırı ekspresyonuna neden olan AmpR (G154R) mutasyonu, OprD (W142X) karbapenem porininde mutasyon, MexXY aşırı ekspresyonuna neden olan MexZ (G195E) mutasyonu ve kinolon direncine neden olan mutasyonlar (GyrA T83I and D87N ve ParC S87W) ile birlikte ÇİD/YİD fenotipi son yıllarda çalışmalarda gösterilmeye başlanmıştır (44). Cabot ve arkadaşlarının (18) yapmış olduğu çalışmada; İspanya’da elde edilen P. aeruginosa izolatının 23’ünde ST175 klonu tanımlanmıştır (YİD, n=19; ÇİD, n=4); dört bölgenin tamamını kapsayan geniş bir coğrafik dağılıma sahip 10 hastanenin yedisinde YİD ST175 izolatı tespit edilmiştir. ST175 yüksek riskli klonuna ait 23 izolatın biri dışında tümü; sefalosporinler, penisilin β-laktamaz inhibitör kombinasyonları, monobaktamlar, karbapenemler, florokinolonlar ve aminoglikozidleri (gentamisin ve tobramisin) içeren benzer bir direnç profiline sahiptir. Tüm ST175 klonuna ait izolatlardaki siprofloksasin direncine neden olan üç florokinolon mutasyonu (GyrA T83I ve D87N ve ParC S87W) saptanmış olup tüm ST175 izolatlarında, gentamisin ve tobramisin direncine yol açan Ant(2”)-Ia’yı kodlayan tek bir gen kaseti olan aadB’yi barındıran sınıf 1 integronu da tespit edilmiştir.
Brezilya’da, bu üç klonun yanı sıra, SPM-1 tipi metalo-β-laktamaz üretimi gösteren ST277 klonu da bir diğer yüksek riskli klondur (35). ST244, her zaman ÇİD/YİD profili göstermemekle birlikte, diğer yüksek riskli klonlara göre daha az sıklıkta tespit edilmektedir (36). Yüksek riskli diğer klonlar arasında yer alan ST308, exoU+ T3SS genotipi ve O11 O-antijen serotipi ile ilişkili olup özellikle IMP-1 tipi karbapenemaz varlığı göstermektedir (46).
İspanya’da bulunan 10 farklı hastanenin katılmış olduğu çok merkezli bir çalışmada, epidemik yüksek riskli klonların başarısıyla potansiyel olarak ilişkili sekiz biyolojik belirteç incelenmiştir. Başlıca üç yüksek riskli klonun (ST235, ST111 ve ST175) üç tip hareketten (seğirme/yüzme/yayılma) ve pigment (piyoverdin ve piyosiyanin) üretiminden yoksun oldukları gösterilmiştir. Öte yandan, bu yüksek riskli klonlar anlamlı şekilde artmış spontan mutasyon sıklığı ve biyofilm üretimi gösteren biyolojik parametreler sergilemektedir (18, 39, 46). Epidemik yüksek riskli klonların başarısına neden olan biyolojik belirteçlerin tam olarak anlaşılması için tüm genom dizilemesine dayalı araştırmalara gereksinim bulunmaktadır.
Kistik Fibrozis Hastalarında Çok İlaca Dirençli P. aeruginosa Epidemik Suşları
Erişkin KF hastalarının %70’i kronik olarak P. aeruginosa ile infekte olup patojen kronik solunum yolu infeksiyonlarından sorumludur. Etkenin erken kazanılması sonucu hastaların kendine ait klonalite göstermeyen ilişkisiz bir suş ile infekte olduğu düşüncesi yerini ÇİD suşların hastalar arasında yayılımı ile ortaya çıkan salgın ile belirli bazı klonların epidemik özellik göstererek dünyada yayıldığı görüşüne bırakmıştır (47). Liverpool epidemik suşu (LES) olarak tanımlanan ST146 klonu, KF hastalarında öne çıkan bir patojendir (48). LES ile kronik infekte olan hastalar kötü prognoza sahip olup bu izolatlar antibiyotik tedavisine daha dirençlidir (49). KF hastalarında LES suşlarının daha yüksek morbidite ve mortaliteye yol açmasının nedeni, bakterinin pyosiyanin ve elastaz A gibi “quorum sensing” ilişkili ekzoürünlerinin aşırı üretimine bağlı gelişen sıra dışı fenotipidir (50). KF hastalarında LES dışında Avustralya epidemik suş (AES)’ları da tanımlanmıştır. AES-1, ST649 klonuna ait olup Melbourne epidemik suşu olarak adlandırılmaktadır (51). Bu epidemik suş dışında, AES-2 ve AES-3 de tanımlanmıştır (52, 53). AES, antibiyotik direncinin ve virulans gen ekspresyonunun fazla olması nedeniyle, KF hastalarının kronik psödomonal infeksiyonlarında yüksek mortalite oranına neden olmaktadır.
P. AERUGINOSA’DA YÜKSEK RİSKLİ KLONLARIN TESPİTİ VE KLİNİK ÖNEMİ
Klinik mikrobiyoloji laboratuvarlarında; ÇİD/YİD yüksek riskli klonların tespiti, epidemiyoloji, infeksiyon kontrolü ve halk sağlığı önlemleri açısından önemlidir. Moleküler mikrobiyolojik yöntemlerin uygulanması bu klonların tespiti açısından değer taşısa da ST235 klonunun saptanmasında basit ve hızlı tarama tekniği olarak O-antijeni serotiplendirmesi öne çıkmaktadır. ST235 için O11, ST111 için O12, ST175 için O4 serotipinin belirlenmesi yüksek riskli klonların saptanmasını sağlamaktadır (54). Bunun dışında ST235, ST175, ST253 ve ST395 gibi yüksek riskli klonların tespitinde MALDI-TOF MS sisteminin (Bruker, ABD) kullanımı doğru ve hızlı tespit açısından değerlidir (55). Soylarının devamını sağlayan sekiz farklı biyolojik parametre incelendiğinde, P. aeruginosa yüksek riskli klonlarının; düşük bakteriyel motilite (yüzme, seğirme, yayılma), siderofor pyoverdin ve pyosiyanin fenazin toksin yapımı ve ortama uyum gücü (rekabet altında üreme) gösterdiği; ancak biyofilm yapımı ile spontan mutasyon hızlarının yüksek olduğu izlenmiştir. Önemli bir virulans belirleyicisi olan Tip III sekresyon sistemi; ExoS, ExoT, ExoU ve ExoT sitotoksinlerini konak hücreye salarak hücrenin ölümüne neden olmaktadır. Bunlardan en potent olan ve ST235 klonunda var olan ExoU kötü prognoz ile ilişkili olup P. aeruginosa infeksiyonlarında prognostik biyobelirteç olma potansiyeli taşımaktadır (56). P. aeruginosa yüksek riskli klonları düşük virulansa sahip görünse de bunlar arasında yer alan ST235 klonu direnç paterninden bağımsız olarak yüksek virulans özellikleri göstermektedir.
SONUÇ
P. aeruginosa rezistomundaki mutasyonel değişiklikler antibiyotiğe maruz kalma ile doğrudan ilişkilidir. Bir antibiyotiğe maruziyet o antibiyotik için P. aeruginosa rezistomunun olumsuz evrilmesine neden olurken, ortaya çıkan bu değişiklik bir diğer antibiyotiğin duyarlılığını olumlu yönde etkileyebilmektedir. Yakın gelecekte özellikle KF ve nozokomiyal P. aeruginosa infeksiyonlarında, P. aeruginosa rezistomunun detaylı analizi yapılarak her hastada doğru tedavi stratejisinin geliştirilmesi sağlanabilecektir. Söz konusu stratejilere uygun olarak başlanan antibiyotik tedavisinin etkinliğinin takip edilmesiyle de çok kullanışlı bir tedavi yaklaşımının ortaya çıkışı mümkün olacaktır.
Danışman Değerlendirmesi
Bağımsız dış danışman.
Yazar Katkıları
Fikir/Kavram – Ö.K.E., Ö.G.; Tasarım – Ö.K.E., Ö.G.; Denetleme – Ö.K.E., Ö.G.; Literatür Taraması – Ö.K.E., Ö.G.; Makale Yazımı – Ö.K.E., Ö.G.; Eleştirel İnceleme – Ö.K.E., Ö.G.
Çıkar Çatışması
Yazarlar herhangi bir çıkar çatışması bildirmemiştir.
Finansal Destek
Yazarlar finansal destek beyan etmemiştir.
Referanslar
- Philip D. Lister, Daniel J. Wolter, Nancy D. Hanson. Antibacterial-resistant Pseudomonas aeruginosa: Clinical impact and complex regulation of chromosomally encoded resistance mechanisms. Clin Microbiol Rev. 2009; 22(4):582-610.
- Aloush V, Navon-Venezia S, Seigman-Igra Y, Cabili S, Carmeli Y. Multidrug-resistant Pseudomonas aeruginosa: risk factors and clinical impact. Antimicrob Agents Chemother. 2006;50(1):43-8. [CrossRef]
- Oliver A, Mulet X, Lopez-Causap. C, Juan C. The increasing threat of Pseudomonas aeruginosa high-risk clones. Drug Resist Updat. 2015;21-22:41-59. [CrossRef]
- Antimicrobial resistance surveillance in Europe 2015 [İnternet]. Stockholm: European Centre for Disease Prevention and Control. [30 Ocak 2017; erişim 01 Eylül 2021]. https://www.ecdc.europa.eu/en/publications-data/antimicrobial-resistance-surveillance-europe-2015
- Tacconelli E, Carrara E, Savoldi A, et al. Discovery, research, and development of new antibiotics: the WHO priority list of antibiotic-resistant bacteria and tuberculosis. Lancet Infect Dis. 2018;18(3):318-27. [CrossRef]
- Alvarez-Ortega C, Wiegand I, Olivares J, Hancock RE, Martínez JL. The intrinsic resistome of Pseudomonas aeruginosa to β-lactams. Virulence. 2011;2(2):144-6. [CrossRef]
- Berrazeg M, Jeannot K, Ntsogo Enguéné VY, et al. Mutations in β-lactamase AmpC increase resistance of Pseudomonas aeruginosa isolates to antipseudomonal cephalosporins. Antimicrob Agents Chemother. 2015;59(10):6248-55. [CrossRef]
- Ramirez MS, Tolmasky ME. Aminoglycoside modifying enzymes. Drug Resist Updat. 2010;13(6):151-71. [CrossRef]
- Alekshun MN, Levy SB. Molecular mechanisms of antibacterial multidrug resistance. Cell. 2007;128(6):1037‐50. [CrossRef]
- Spengler G, Kincses A, Gajdács M, Amaral L. New roads leading to old destinations: efflux pumps as targets to reverse multidrug resistance in bacteria. Molecules. 2017;22(3):468. [CrossRef]
- Li XZ, Plésiat P, Nikaido H. The challenge of efflux-mediated antibiotic resistance in Gram-negative bacteria. Clin Microbiol Rev. 2015;28(2): 337-418. [CrossRef]
- Dupont P, Hocquet D, Jeannot K, Chavanet P, Plésiat P. Bacteriostatic and bactericidal activities of eight fluoroquinolones against MexAB-OprM-overproducing clinical strains of Pseudomonas aeruginosa. J Antimicrob Chemother. 2005;55(4):518-22. [CrossRef]
- Okamoto K, Gotoh N, Nishino T. Extrusion of penem antibiotics by multicomponent efflux systems MexAB-OprM, MexCD-OprJ, and MexXY-OprM of Pseudomonas aeruginosa. Antimicrob Agents Chemother. 2002;46(8):2696-9. [CrossRef]
- Llanes C, Köhler T, Patry I, Dehecq B, Van Delden C, Plésiat P. Role of the MexEF-OprN efflux system in low-level resistance of Pseudomonas aeruginosa to ciprofloxacin. Antimicrob Agents Chemother. 2011;55(12):5676-84. [CrossRef]
- Chevalier S, Bouffartigues E, Bodilis J, et al. Structure, function and regulation of Pseudomonas aeruginosa porins. FEMS Microbiol Rev. 2017;41(5):698-722. [CrossRef]
- Moya B, Dötsch A, Juan C, et al. Beta-lactam resistance response triggered by inactivation of a nonessential penicillin-binding protein. PLoS Pathog. 2009;5(3):e1000353. [CrossRef]
- Juan C, Maci MD, Gutirrez O, Vidal C, Perez JL, Oliver A. Molecular mechanisms of beta-lactam resistance mediated by AmpC hyperproduction in Pseudomonas aeruginosa clinical strains. Antimicrob Agents Chemother. 2005;49(11):4733-8. [CrossRef]
- Cabot G, Ocampo-Sosa AA, Tubau F, et al. Overexpression of AmpC and efflux pumps in Pseudomonas aeruginosa isolates from bloodstream infections: prevalence and impact on resistance in a Spanish multicenter study. Antimicrob Agents Chemother. 2011;55(5):1906-11. [CrossRef]
- Fraile-Ribot PA, Cabot G, Mulet X, et al. Mechanisms leading to in vivo ceftolozane/ tazobactam resistance development during the treatment of infections caused by MDR Pseudomonas aeruginosa. J Antimicrob Chemother. 2018;73(3):658-63. [CrossRef]
- Pseudomonas aeruginosa Derived Cephalosporinase (PDC) Database [Internet]. Palma: Antibiotic Resistance and Pathogenicity of Bacterial Infections Group. [güncelleme 10 Aralık 2021; erişim 01 Eylül 2021]. https://arpbigidisba.com
- Cabot G, Florit-Mendoza L, Sanchez-Diener I, Zamorano L, Oliver A. Deciphering beta-lactamase-independent beta-lactam resistance evolution trajectories in Pseudomonas aeruginosa. J Antimicrob Chemother. 2018;73(12):3322-31. [CrossRef]
- Riera E, Cabot G, Mulet X, et al. Pseudomonas aeruginosa carbapenem resistance mechanisms in Spain: impact on the activity of imipenem, meropenem and doripenem. J Antimicrob Chemother. 2011;66(9):2022-7. [CrossRef]
- Moy B, Beceiro A, Cabot G, et al. Pan-beta-lactam resistance development in Pseudomonas aeruginosa clinical strains: molecular mechanisms, penicillin-binding protein profiles, and binding affinities. Antimicrob Agents Chemother. 2012;56(9):4771-8. [CrossRef]
- Guenard S, Muller C, Monlezun L, et al. Multiple mutations lead to MexXY-OprM-dependent aminoglycoside resistance in clinical strains of Pseudomonas aeruginosa. Antimicrob Agents Chemother. 2014;58(1):221-8. [CrossRef]
- Köhler T, Epp SF, Curty LK, Pechère JC. Characterization of MexT, the regulator of the MexE-MexF-OprN multidrug efflux system of Pseudomonas aeruginosa. J Bacteriol. 1999;181(20):6300–5. [CrossRef]
- Mulet X, Moy B, Juan C, et al. Antagonistic interactions of Pseudomonas aeruginosa antibiotic resistance mechanisms in planktonic but not biofilm growth. Antimicrob Agents Chemother. 2011;55(10):4560-8. [CrossRef]
- Bruchmann S, Dötsch A, Nouri B, Chaberny IF, Häussler S. Quantitative contributions of target alteration and decreased drug accumulation to Pseudomonas aeruginosa fluoroquinolone resistance. Antimicrob Agents Chemother. 2013;57(3):1361-8. [CrossRef]
- Del Barrio-Tofiño E, López-Causapé C, Cabot G, et al. Genomics and susceptibility profiles of extensively drug-resistant Pseudomonas aeruginosa isolates from Spain. Antimicrob Agents Chemother. 2017;61(11):e01589-17. [CrossRef]
- López-Causapé C, Cabot G, Del Barrio-Tofiño E, Oliver A. The versatile mutational resistome of Pseudomonas aeruginosa. Front Microbiol. 2018;9:685. [CrossRef]
- Dößelmann, B, Willmann M, Steglich M. Rapid and consistent evolution of colistin resistance in extensively drugresistant Pseudomonas aeruginosa during morbidostat culture. Antimicrob Agents Chemother. 2017;61(9):e00043-17. [CrossRef]
- Patel G, Bonomo RA. Status report on carbapenemases: challenges and prospects. Expert Rev Anti Infect Ther. 2011;9(5):555-70. [CrossRef]
- Diene SMM, Rolain JM. Carbapenemase genes and genetic platforms in Gram-negative bacilli: Enterobacteriaceae, Pseudomonas and Acinetobacter species. Clin Microbiol Infect. 2014;20(9):831-8. [CrossRef]
- Chávez-Jacobo VM, Hernández-Ramírez KC, Romo-Rodríguez P, et al. CrpP Is a novel ciprofloxacin-modifying enzyme encoded by the Pseudomonas aeruginosa pUM505 plasmid. Antimicrob Agents Chemother. 2018;62(6):e02629-17. [CrossRef]
- Pál C, Papp B, Lázár V. Collateral sensitivity of antibiotic-resistant microbes. Trends Microbiol. 2015;23(7):401-7. [CrossRef]
- Imamovic L, Ellabaan MMH, Dantas Machado AM, et al. Drug-driven phenotypic convergence supports rational treatment strategies of chronic infections. Cell. 2018;172(1-2):121-134.e14. [CrossRef]
- Cabot G, Bruchmann S, Mulet X. Pseudomonas aeruginosa ceftolozane-tazobactam resistance development requires multiple mutations leading to overexpression and structural modification of AmpC. Antimicrob Agents Chemother. 2014;58(6):3091-9. [CrossRef]
- Yen P, Papin JA. History of antibiotic adaptation influences microbial evolutionary dynamics during subsequent treatment. PLoS Biol. 2017;15(8):e2001586. [CrossRef]
- Del Barrio-Tofiño E, López-Causapé C, Oliver A. Pseudomonas aeruginosa epidemic high-risk clones and their association with horizontally-acquired β-lactamases: 2020 update. Int J Antimicrob Agents. 2020;56(6):106196. [CrossRef]
- Cabot G, Ocampo-Sosa AA, Domínguez MA. Genetic markers of widespread extensively drug-resistant Pseudomonas aeruginosa high-risk clones. Antimicrob Agents Chemother. 2021;56(12):6349-57. [CrossRef]
- Thrane SW, Taylor VL, Freschi L, et al. The widespread multidrug-resistant serotype O12 Pseudomonas aeruginosa clone emerged through concomitant horizontal transfer of serotype antigen and antibiotic resistance gene clusters. mBio. 2015;6(5):e01315–96. [CrossRef]
- Treepong P, Kos VN, Guyeux C, et al. Global emergence of the widespread Pseudomonas aeruginosa ST235 clone. Clin Microbiol Infect. 2018;24(3):258-66. [CrossRef]
- Kocsis B, Gulyás D, Szabó D. Diversity and distribution of resistance markers in Pseudomonas aeruginosa international high-risk clones. Microorganisms. 2021;9(2):359. [CrossRef]
- Turton JF, Wright L, Underwood A, et al. High-resolution analysis by whole-genome sequencing of an international lineage (Sequence Type 111) of Pseudomonas aeruginosa associated with metallo-carbapenemases in the United Kingdom. J Clin Microbiol. 2015;53(8):2622-31. [CrossRef]
- Cabot G, López-Causapé C, Ocampo-Sosa AA, et al. Deciphering the resistome of the widespread Pseudomonas aeruginosa sequence type 175 international high-risk clone through wholegenome sequencing. Antimicrob Agents Chemother. 2016;60(12):7415-23. [CrossRef]
- Fraile-Ribot PA, Del Rosario-Quintana C, López-Causapé C, et al. Emergence of resistance to novel β-lactam–β-lactamase inhibitor combinations due to horizontally acquired AmpC (FOX-4) in Pseudomonas aeruginosa sequence type 308. Antimicrob Agents Chemother. 2019;64(1):e02112-19. [CrossRef]
- Mulet X, Cabot G, Ocampo-Sosa AA, et al. Biological markers of Pseudomonas aeruginosa epidemic high-risk clones. Antimicrob Agents Chemother. 2013;57(11): 5527-35. [CrossRef]
- Pedersen SS, Koch C, Høiby N, Rosendal K. An epidemic spread of multiresistant Pseudomonas aeruginosa in a cystic fibrosis centre. J Antimicrob Chemother. 1986;17(4):505-16. [CrossRef]
- López-Causapé C, Rojo-Molinero E, Mulet X, et al. Clonal dissemination, emergence of mutator lineages and antibiotic resistance evolution in Pseudomonas aeruginosa cystic fibrosis chronic lung infection. PLoS ONE. 2013;8(8):e71001. [CrossRef]
- Ashish A, Shaw M, McShane J, Ledson MJ, Walshaw MJ. Health-related quality of life in cystic fibrosis patients infected with transmissible Pseudomonas aeruginosa strains: cohort study. JRSM Short Rep. 2012;3(2):12. [CrossRef]
- Fothergill JL, Panagea S, Hart CA, Walshaw MJ, Pitt TL, Winstanley C. Widespread pyocyanin over-production among isolates of a cystic fibrosis epidemic strain. BMC Microbiol. 2007;7:45. [CrossRef]
- Armstrong D, Bell S, Robinson M, et al. Evidence for spread of a clonal strain of Pseudomonas aeruginosa among cystic fibrosis clinics. J Clin Microbiol. 2003;41(5):2266-7. [CrossRef]
- Anthony M, Rose B, Pegler MB, et al. Genetic analysis of Pseudomonas aeruginosa isolates from the sputa of Australian adult cystic fibrosis patients. J Clin Microbiol. 2002;40(8):2772-8. [CrossRef]
- Bradbury R, Champion A, Reid DW. Poor clinical outcomes associated with a multi-drug resistant clonal strain of Pseudomonas aeruginosa in the Tasmanian cystic fibrosis population. Respirology. 2008;13(6):886-92. [CrossRef]
- Del Barrio-Tofiño E, Sánchez-Diener I, Zamorano L, et al. Association between Pseudomonas aeruginosa O-antigen serotypes, resistance profiles and high-risk clones: results from a Spanish nationwide survey. J Antimicrob Chemother. 2019;74(11):3217-20. [CrossRef]
- Cabrolier, N., Sauget, M., Bertrand, X., Hocquet, D. Matrix-assisted laser desorption ionization-time of flight mass spectrometry identifies Pseudomonas aeruginosa high-risk clones. J Clin Microbiol. 2015;53(4):1395-8. [CrossRef]
- Agnello M, Finkel SE, Wong-Beringer A. Fitness cost of fluoroquinolone resistance in clinical isolates of Pseudomonas aeruginosa differs by Type III secretion genotype. Front Microbiol. 2016;7:1591. [CrossRef]