En Çok Okunanlar
Özet
Amaç: Son yıllarda, toplum ve hastane kaynaklı infeksiyonlarda en sık görülen etkenlerden biri olan enterokoklarda giderek artan oranda çoklu antibiyotik direnci gözlenmektedir. Özellikle vankomisine dirençli enterokokların tedavisi ciddi sorun oluşturmaktadır. Günümüzde Enterococcus türlerinin tanımlanmasında ve antibiyotik dirençlerinin belirlenmesinde konvansiyonel kültür testlerine dayalı fenotipik yöntemlerin yanı sıra, tür ve direnç genlerinin polimeraz zincir reaksiyonu (PCR) ile genotipik olarak tanımlanması da kullanılmaktadır. Son yıllarda, aynı reaksiyonda birden fazla genin çoğaltılabildiği multipleks PCR (mPCR) yöntemi, tür ve antibiyotik direnç genlerinin daha hızlı ve ekonomik olarak tanımlanmasına olanak sağlamaktadır. Bu çalışmada, Enterococcus izolatlarında tür ve vankomisin direnç tespitinde mPCR yöntemi ile konvansiyonel kültür test sonuçlarının karşılaştırılması amaçlandı.
Yöntemler: Bu çalışmada, 2015–2019 yılları arasında farklı klinik örneklerinden izole edilmiş, konvansiyonel yöntemlerle tanımlanarak saklanan 38 Enterococcus izolatının tür ve vankomisin direnç profilleri genotipik olarak mPCR yöntemi ile araştırıldı.
Bulgular: Kültür ve moleküler tanı testlerinde, 38 izolatın 31’inde tür ve direnç bakımından %82 oranında uyum saptandı. Çalışmaya dahil edilen izolatlar arasında vanA dirençli Enterococcus faecium’un baskın tür olduğu belirlendi. Olağandışı olarak, bir Enterococcus faecalis izolatının bu türde yaygın olan vanB yerine vanA geni taşıdığı ve bir Enterococcus gallinarum izolatının da vanC-1 ve vanA tipi vankomisin direnç genlerini birlikte taşıdığı tespit edildi.
Sonuçlar: Bu çalışma, mPCR yönteminin etkinliğini ve vankomisin dirençli enterokokların genotipik profilindeki çeşitliliği ortaya koymaktadır. Klinik izolatlarda nadir görülen horizontal gen transferi olasılığı, direnç yayılımı açısından önemli klinik ve epidemiyolojik çıkarımlar sunmaktadır. Vankomisine dirençli enterokokların yayılımını engelleyecek önlemler alınması önem taşımaktadır.
GİRİŞ
Gerek toplum gerekse hastane kökenli infeksiyonlara neden olan patojen bakterilerin tedavisinde antibiyotik direnci, dünyada ve ülkemizde giderek artan bir sorundur (1). Enterococcus türleri insan ve hayvanların sindirim sisteminin normal florasında bulunan ancak bakteriyemi, endokardit, üriner sistem infeksiyonları, intraabdominal ve pelvik infeksiyonlar, yara ve doku infeksiyonları, kolesistit, menenjit, neonatal sepsis, hastane kaynaklı pnömoni ve septisemi gibi infeksiyonlara da neden olabilen fırsatçı patojen bakterilerdir (2).
Çoklu antibiyotik direnci gösteren Enterococcus izolatlarının ortaya çıkması ile mevcut tedavi yöntemlerinin uygulanabilirliği kısıtlanmış; glikopeptid, penisilin ve aminoglikozit grubu antibiyotiklere direnç genleri taşıyan enterokok infeksiyonları halk sağlığı açısından önemli bir tehdit haline gelmiştir (3). Vankomisine dirençli enterokok (Vancomycin-resistant Enterococcus, VRE) suşları, infekte veya kolonize hastalar, hastane personeli ve çevre kontaminasyonu aracılığıyla yayılmakta; salgınlara ve tedavisi güç infeksiyonlara neden olabilmektedir. Bu nedenle, klinik örnekler, rektal sürüntüler ve çevre örneklerinde VRE suşlarının araştırılması, yayılımın önlenmesi açısından önem taşımaktadır (4).
Glikopeptid grubu antibiyotikler (vankomisin, teikoplanin) enterokoklarda peptidoglikan sentezini, dolayısıyla hücre duvarı sentezini engelleyerek etki gösterir. Hücre duvarının ana bileşeni olan peptidoglikan sentezi sırasında iki D-alanin molekülünün birbirine bağlanması, bir ligaz enzimi (ddl) tarafından katalize edilir. Oluşan D-alanin-D-alanin dipeptidi, devam eden enzimatik reaksiyonlarla UDP-N-asetilmuramil-tripeptide eklenir ve UDP-N-asetilmuramil-pentapeptidi oluşturulur. Bu pentapeptit molekülü, transglikolizasyon yoluyla sitoplazmik membranın dış yüzeyine taşınarak peptidoglikan sentezine katılır. Pentapeptidin serbest karboksil ucunda bulunan terminal D-alanin-D-alanin bölgesine yüksek afinite ile bağlanan vankomisin, peptidoglikan sentezinin transglikolizasyon basamağını inhibe ederek hücre duvarı sentezini durdurur ve bakterisidal etki oluşturur.
Enterokoklarda vankomisin direnci, düşük afiniteli pentapeptit öncüllerinin sentezlenmesinden kaynaklanır. Vankomisine dirençli enterokok suşları farklı bir ligaz enzimi sentezleyerek D-alanin-D-alanin’deki alanin yerine laktat veya serin bağlar; bu değişiklik uç kısmın yapısını değiştirerek vankomisinin hedef bölgeye bağlanma yeteneğini azaltır. Böylece D-alanin-D-laktat veya D-alanin-D-serin içeren modifiye pentapeptidler vankomisinin hücre duvarına bağlanma yeteneğinin azalmasına neden olur.
Enterokoklarda iki tip vankomisin direnci görülür. Birincisi, kromozom üzerinde yerleşik genlere bağlı doğal (intrensek) dirençtir. Modifiye D-alanin-D-serin taşıyan Enterococcus gallinarum ve Enterococcus casseliflavus izolatları vankomisine doğal fakat düşük düzeyli bir direnç gösterir. İkinci tip direnç ise başka bir organizmadan genetik transferle kazanılmış (ekstrensek) dirençtir. Bu direnç tipi en sık modifiye D-alanin-D-laktat taşıyan Enterococcus faecium ve Enterococcus faecalis izolatlarında görülür (5).
Bugüne kadar enterokoklarda klinik açıdan farklı öneme sahip dokuz vankomisin direnç tipi (vanA, vanB, vanC, vanD, vanE, vanG, vanL, vanM ve vanN) tanımlanmıştır. Klinik olguların çoğunu oluşturan E. faecium ve E. faecalis türlerinde sırasıyla VanA ve VanB direncinin ve daha az görülen E. gallinarum türünde ise VanC-1 direncinin yaygın olduğu bildirilmiştir (6).
Enterokok izolatlarının tür ve vankomisin direnç tespitinde, konvansiyonel ve kültüre dayalı fenotipik testlerin yanı sıra, tür ve direnç genlerinin hızlı ve doğru biçimde belirlenmesine olanak sağlayan moleküler teknikler de geliştirilmiştir. Bu teknikler arasında türe özgü değişkenlik gösteren D-alanin-D-alanin ligazlarını kodlayan ddl genlerinin ve vankomisin direnç genlerinin polimeraz zincir reaksiyonu (PCR) ile amplifikasyonu yer almaktadır (7-10).
Bu çalışmada, hızlı ve pratik olan multipleks-PCR (mPCR) yöntemi kullanılarak hastanenin servis ve yoğun bakım ünitelerindeki hastaların çeşitli klinik örneklerinden izole edilen 38 Enterococcus izolatının tür ve direnç profillerinin genotipik olarak belirlenmesi ve bulguların konvansiyonel kültür test sonuçlarıyla karşılaştırılması planlandı. Bu amaçla, klinik enterokok izolatlarında en sık görülen E. faecium, E. faecalis ve E. gallinarum türlerinin dağılımı ile vanA, vanB ve vanC-1 direnç genlerinin varlığı, tek reaksiyonda ve yüksek duyarlılıkla tespit edebilen optimize bir mPCR protokolü ile araştırıldı (11).
YÖNTEMLER
Klinik İzolatlar ve Referans Suşlar
2015–2019 yılları arasında hastanenin servis ve yoğun bakım ünitelerinde yatan hastalardan alınan klinik örneklerden Tıbbi Mikrobiyoloji Laboratuvarına’nda izole edilip kültür koleksiyonunda saklanan 38 Enterococcus izolatı çalışmaya dahil edildi.
Klinik örnekler koyun kanlı agar ve “eosin methylene blue” (EMB) agara ekilerek 37˚C’de 24–48 saat inkübe edildi. İnkübasyon sonrası kanlı agarda uygun koloni morfolojisine sahip örneklere Gram boyaması, katalaz testi, L-pirolidonil-β-naftilamid testi (PYR) testi gibi rutin laboratuvar yöntemleri uygulandı.
Enterokok şüphesi bulunan suşların tür düzeyinde tanımlanması ve antibiyotik duyarlılık testleri VITEK® 2 (bioMérieux, Marcy l’Etoile, Fransa) otomatize sistemi ile yapıldı. Otomatize sistemde vankomisin dirençli bulunan izolatlarda, Avrupa Antimikrobiyal Duyarlılık Testi Komitesi (European Committee on Antimicrobial Susceptibility Testing, EUCAST) önerileri doğrultusunda Mueller-Hinton agar (MHA) besiyeri kullanılarak vankomisin (5µg) diski ile Kirby-Bauer disk difüzyon testi uygulandı (12). Referans suşlar (VanA dirençli E. faecium, VanB dirençli E. faecalis ve VanC-1 dirençli E. gallinarum) Türkiye Halk Sağlığı Kurumu Moleküler Mikrobiyoloji Araştırma Laboratuvarı’ndan temin edilerek, tür ve vankomisin direnç genleri için pozitif kontrol olarak kullanıldı.
Bu çalışma için Ahi Evran Üniversitesi Girişimsel Olmayan Klinik Araştırmalar Etik Kurulu’ndan 20 Ekim 2020 tarih ve 2020-15/106 karar numarasıyla onay alındı.
DNA İzolasyonu
Moleküler analizlerde kullanılacak DNA’nın elde edilmesi için -80°C’de saklanan bakteri izolatları katı besiyerinde üretilerek canlandırıldı. Referans ve klinik Enterococcus izolatların total DNA’sı kaynatma yöntemiyle elde edildi (13). Elde edilen DNA örnekleri kullanılıncaya kadar -20°C’de saklandı.
Kullanılan Primerler
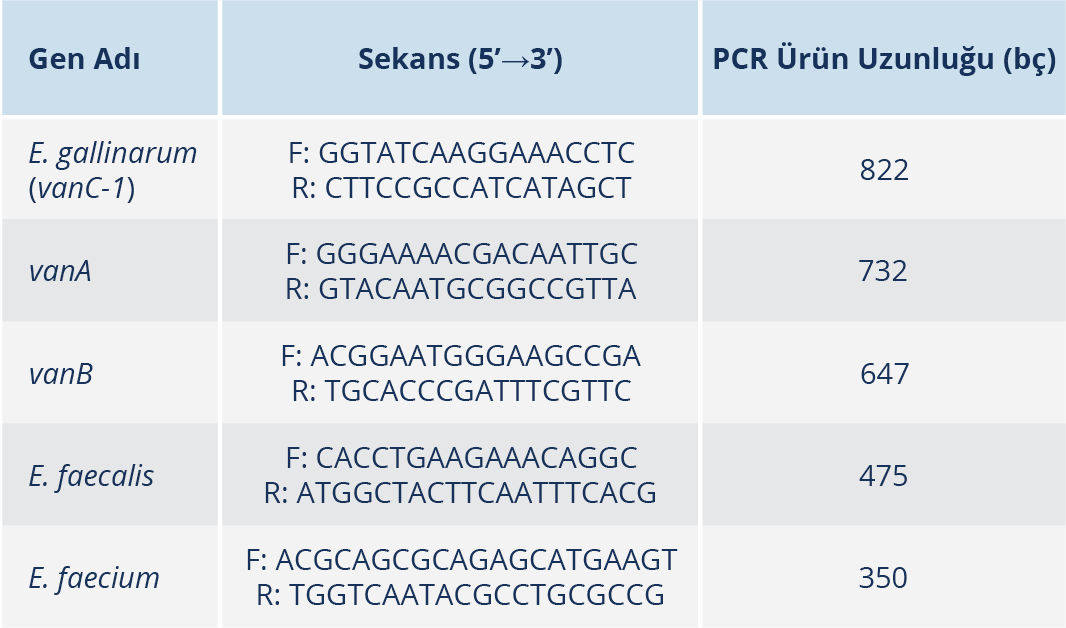
Çalışmada hedeflenen E. faecium, E. faecalis, E. gallinarum (vanC-1), vanA ve vanB genlerine özgü beş çift primer kullanıldı. Primer dizileri ve beklenen amplifikasyon ürünlerinin uzunlukları (baz çifti, bç) Tablo 1’de sunuldu.
mPCR ile Tür ve Vankomisin Direnç Genlerinin Amplifikasyonu
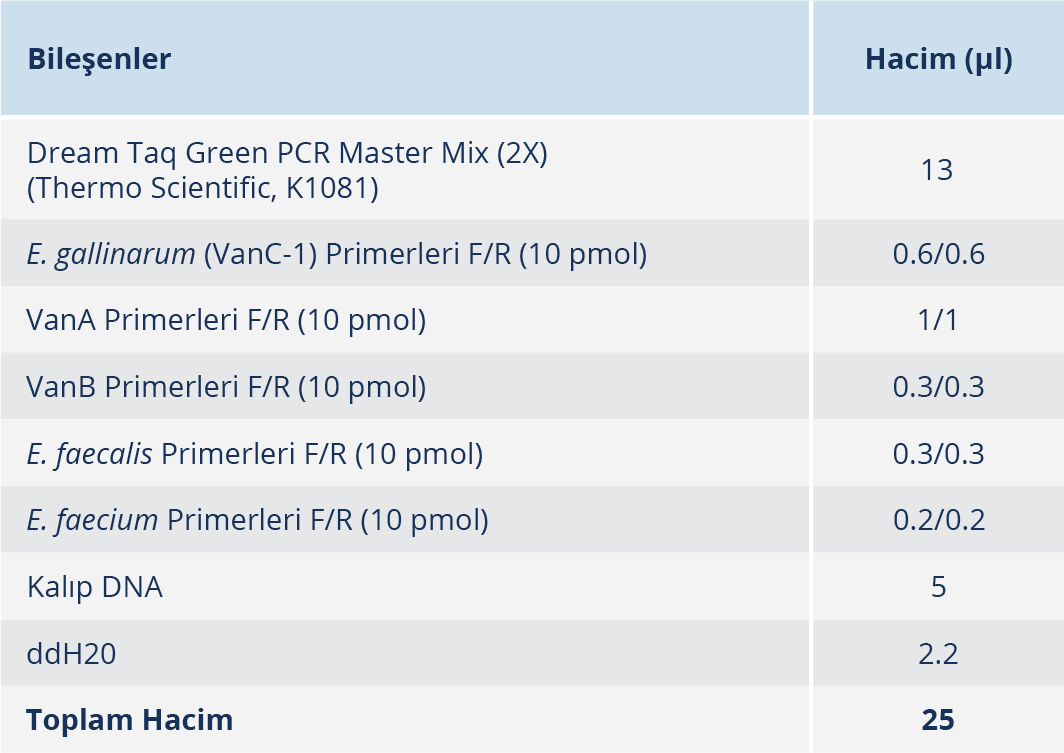
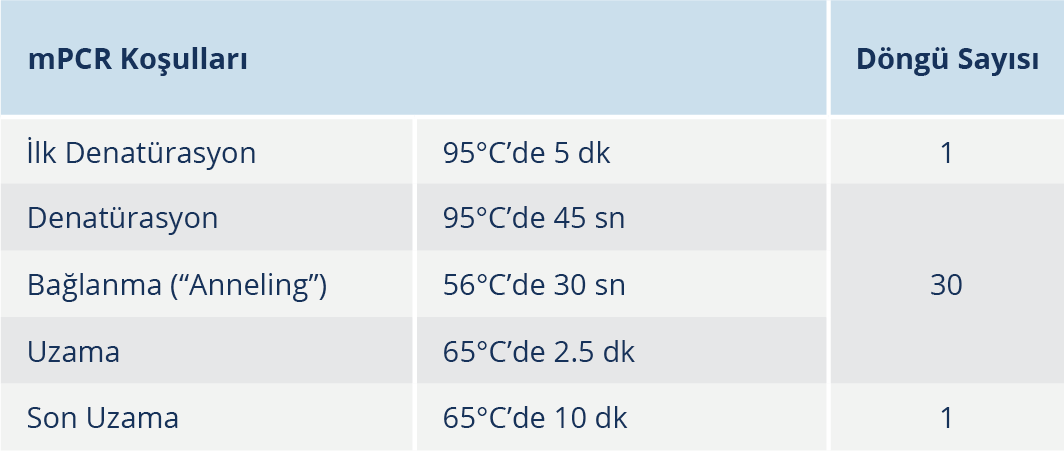
Tür ve vankomisin direnç genlerinin tek reaksiyonda çoğaltılmasını sağlayan optimize mPCR karışımının içeriği Tablo 2’de, amplifikasyon koşulları ise Tablo 3’te sunuldu (11).
Tablo 2’de belirtilen tüm bileşenleri içeren ancak kalıp DNA eklenmemiş “master mPCR reaksiyon karışımı” referans suşlar ve test edilecek klinik izolatların sayısı kadar hazırlandı. Referans suşlar ve test edilecek izolatlar için ayrı ayrı hazırlanan 0.2 ml’lik PCR tüplerine bu karışımdan 20’şer µl dağıtıldıktan sonra 5’er µl kalıp DNA ilave edildi. Tüpler vorteksle karıştırıldıktan sonraT100™ Thermal Cycler (Bio-Rad Laboratories, Hercules, CA, ABD) cihazında amplifikasyon işlemi gerçekleştirildi.
Agaroz Jel Elektroforezi
Referans suşlar ve klinik izolatlardan elde edilen mPCR ürünleri %2’lik agaroz jelde elektroforezle ayrıştırıldı. DNA bantları, Gel Doc™ XR görüntüleme sistemiyle (Bio-Rad Laboratories, Hercules, CA, ABD) görüntülendi. Elde edilen amplikon büyüklükleri, 100 bç’lik DNA belirteci bantlarıyla karşılaştırılarak tür ve direnç genlerinin varlığı değerlendirildi.
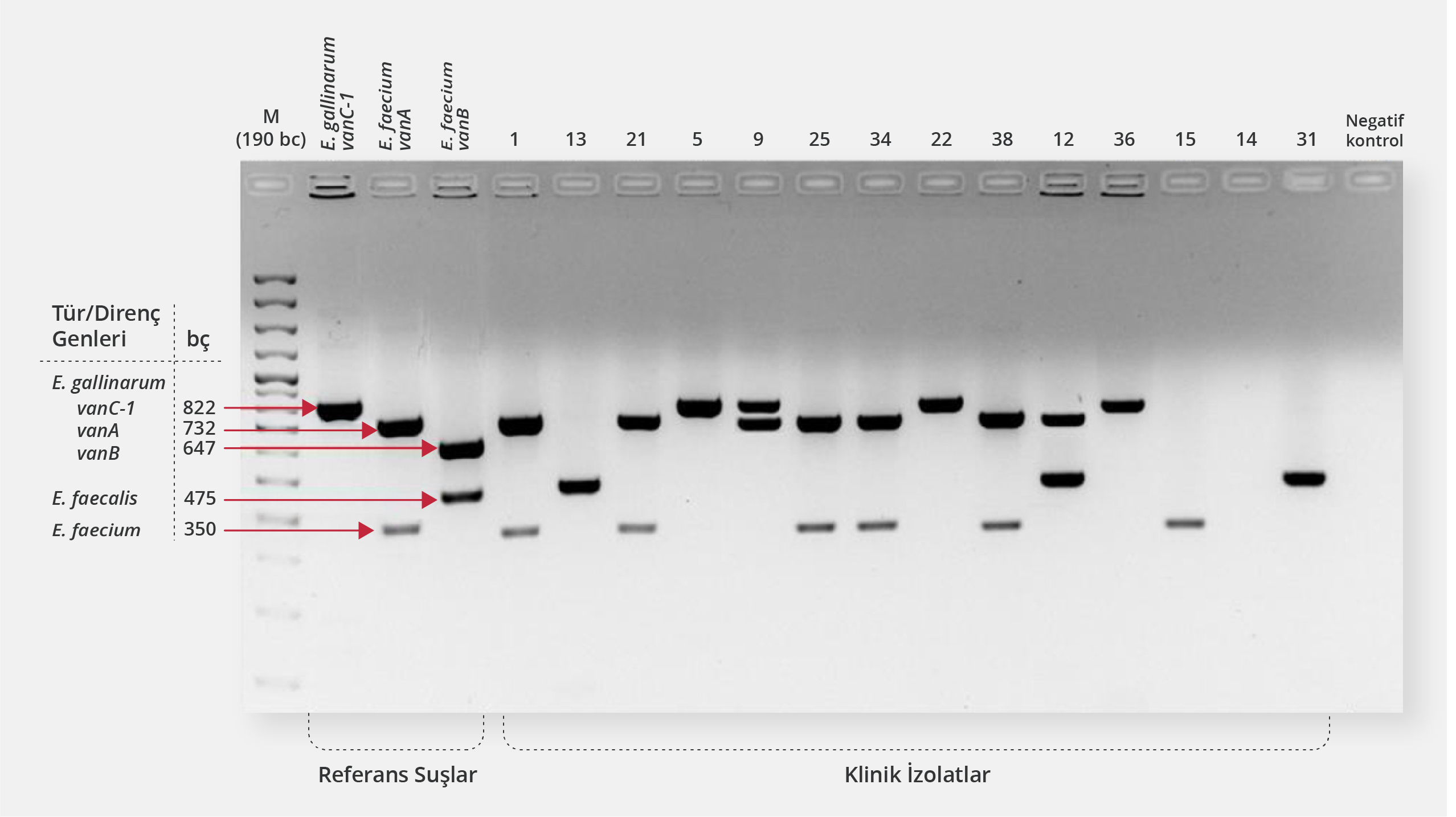
Şekil 1. Referans suşlar ve klinik izolatların örnek agaroz jel elektroforez görüntüsü.
1; E. faecium (vanA), 13; E. faecalis, 21; E. faecium (vanA), 5; E. gallinarum (vanC-1), 9; E. gallinarum (vanC-1 ve vanA), 25; E. faecium (vanA), 34; E. faecium (vanA), 22; E. gallinarum (vanC-1), 38; E. faecium (vanA), 12; E. faecalis (vanA), 36; E. gallinarum (vanC-1), 15; E. faecium, 14; türü tespit edilemeyen, 31; E. faecalis.
Klinik örneklerde saptanan amplifikasyon ürünleri, referans suşlara ait bantlar ve DNA belirteç bantlarıyla karşılaştırıldı. Şekil 1’de verilen örnek jel elektroforez resminde, pozitif kontrol olarak kullanılan referans suşlarda tür ve direnç genlerine özgü beş amplikon belirlendi. DNA içermeyen negatif kontrol örneğinde beklendiği üzere herhangi bir amplifikasyon ürünü gözlenmedi.
Aynı jel üzerinde analiz edilen 14 klinik izolatın tür ve direnç profilleri, amplifikasyon bantlarının varlığına göre değerlendirildi (Şekil 1). Tüm klinik örnekler aynı şekilde analiz edilerek moleküler karakterizasyonları gerçekleştirildi. Deneyler her örnek için iki kez tekrarlandı.
BULGULAR
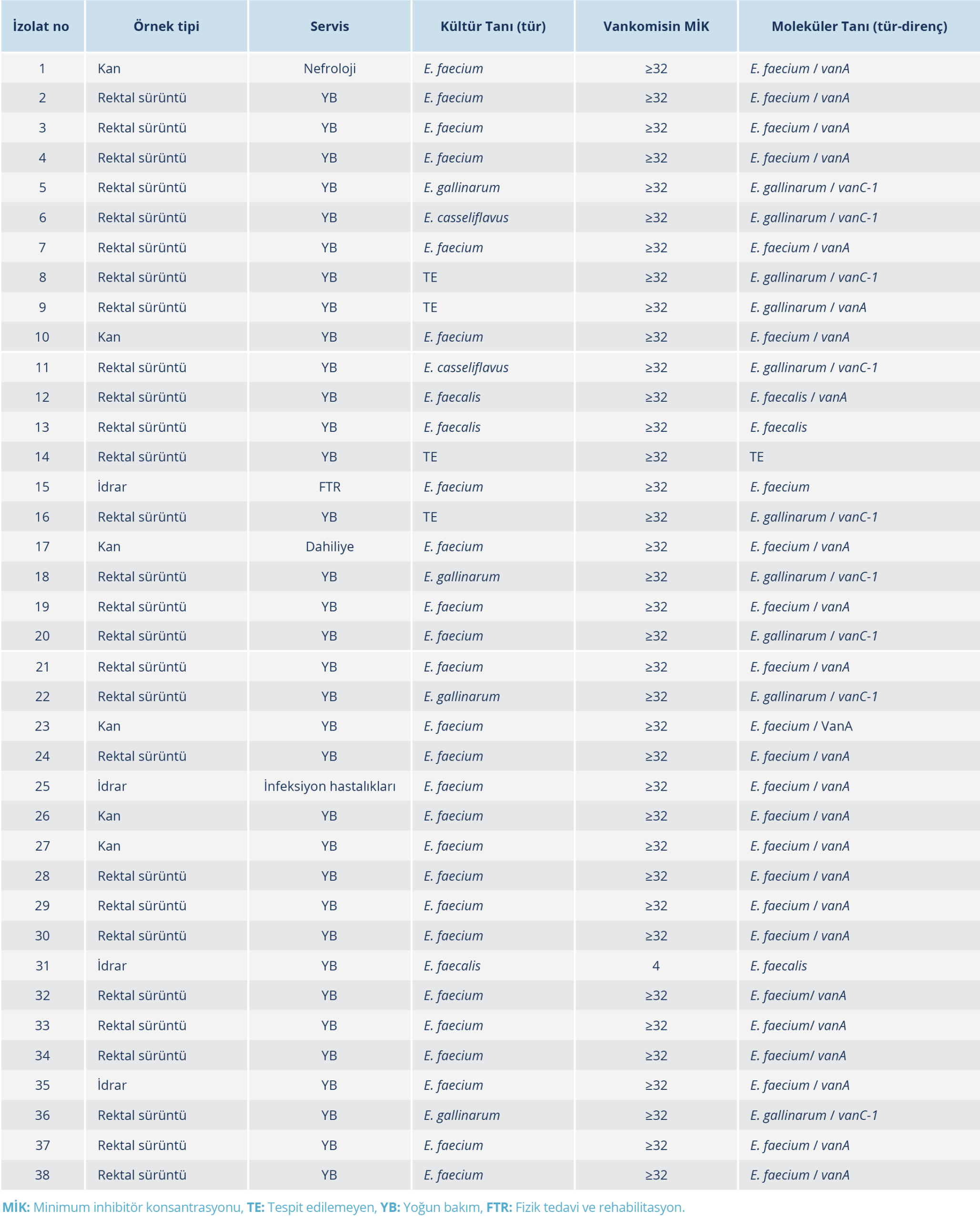
Çalışmada incelenen 38 örneğin 28’i tarama örneklerinden, 10’u klinik örneklerden elde edildi. Bu izolatlara ait bilgiler ve kültür yöntemi ile elde edilen tür ve direnç profilleri Tablo 4’te sunuldu. İzolatların 34’ü yoğun bakım ünitesinde yatan hastalara aitti; 28’i rektal sürüntü, 4’ü kan ve 2’si ise idrar örneklerinden elde edilmişti. Geriye kalan 4 örneğin 2’si kan, 2’si idrar örneği olup sırasıyla dahiliye, nefroloji, infeksiyon hastalıkları ve fizik tedavi ve rehabilitasyon servislerin gönderilmişti.
mPCR Analizi
Çalışmamızda, mPCR sonuçlarına göre klinik izolatların 8’i VanA dirençli E. faecium, 1’i duyarlı E. faecium ve 1’i ise duyarlı E. faecalis olarak tanımlandı. Çoğunluğu oluşturan 28 yoğun bakım rektal sürüntü örneğinin 15’i VanA dirençli E. faecium, 9’u VanC-1 dirençli E. gallinarum, 1’i hem VanC-1 hem de VanA dirençli E. gallinarum, 1’i duyarlı E. faecalis ve 1’i VanA dirençli E. faecalis idi; rektal sürüntü örneklerinden yalnızca 1’nde tür ve direnç tanımlaması yapılamadı (Tablo 4).
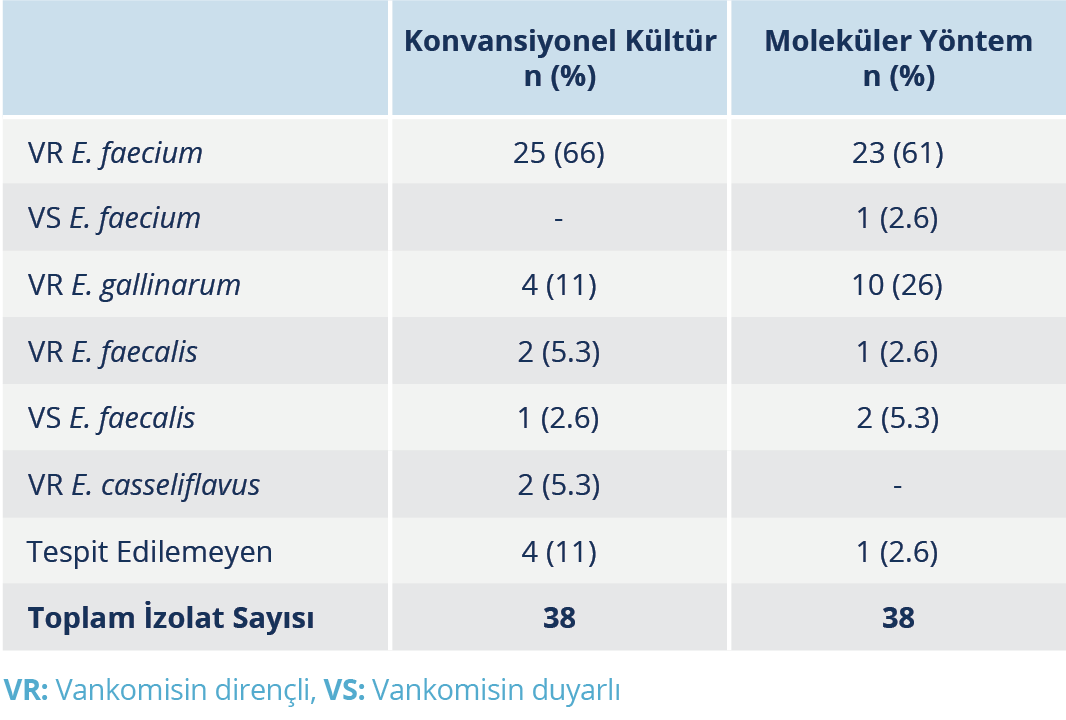
Laboratuvarımızda konvansiyonel kültür testleriyle elde edilen sonuçlara göre, 38 izolatın dağılımı şu şekilde idi: 25 (%66) vankomisin dirençli E. faecium, 4 (%11) vankomisin dirençli E. gallinarum, 2 (%5.3) vankomisin dirençli E. faecalis, 1 (%2.6) duyarlı E. faecalis, 2 (%5.3) vankomisin dirençli E. casseliflavus ve 4 (%11) türü tespit edilemeyen izolat.
mPCR sonuçlarına göre ise 38 izolatın tür ve direnç profilleri şu şekilde bulundu: 23 (%61) vankomisin dirençli E. faecium, 1 (%2.6) duyarlı E. faecium, 10 (%26) vankomisin dirençli E. gallinarum, 1 (%2.6) vankomisin dirençli E. faecalis, 2 (%5.3) duyarlı E. faecalis ve sadece 1 (%2.6) türü tespit edilemeyen izolat (Tablo 5).
Kültür ve mPCR yöntemleri karşılaştırıldığında, 38 izolatın 31’inde tür ve direnç açısından tam uyum saptanmış olup iki yöntem arasındaki uyum oranı %82, tanısal farklılık oranı ise %18 olarak tespit edildi.
İRDELEME
Vankomisine dirençli enterokoklar 1980’lerde Avrupa’da ve 1993’te Kanada’da tanımlanmış; kısa sürede dünya genelinde ciddi hastalıklara neden olan en önemli hastane kaynaklı patojenler arasına girmiştir (14). Günümüzde gerek hastane gerekse toplum kökenli Enterococcus izolatlarında vankomisin direncinin oldukça yüksek düzeylere ulaştığı bildirilmektedir ve vankomisin direncinden sorumlu genlerin tespiti ve prevalansının takibi hastanelerde direnç yayılımının önlenmesi açısından son derece önemlidir (2).
Enterococcus türlerinde vankomisine direnç gelişimine katkıda bulunan dokuz farklı tipte direnç operonu (vanA, vanB, vanC, vanD, vanE, vanG, vanL, vanM ve vanN) tanımlanmıştır. Bunlar arasında VanA ve VanB kaynaklı direnç klinik olarak en önemlileridir. VanA tipi direnç hem vankomisine hem de teikoplanine yüksek düzeyde direnç kazandırırken, VanB tipi direnç yalnızca vankomisine karşı etkilidir. Hem vanA hem de vanB gen kümeleri sırasıyla Tn1546 ve Tn1549 mobil elementleri (transpozonlar) üzerinde yerleşik olduğundan vanA ve vanB aracılı vankomisin direnci doğrudan bu mobil elementler ya da bunları taşıyan plazmidlerin izolatlar arasında transferi ile yayılmaktadır (15). Buna karşılık, kromozomal lokasyonu nedeniyle aktarılamayan bir operon olan vanC gen kümesi E. gallinarum ve E. casseliflavus izolatlarında vankomisine düşük düzeyde direnç oluşturur (16).
İnsan klinik örneklerinden izole edilen VRE’ler arasında en sık rastlanan tür VanA tipi dirençli E. faecium’dur; bunu VanB dirençli E. faecalis izlemektedir. Klinik örneklerde daha seyrek görülen ve bu nedenle önemsiz olduğu düşünülen VanC grubu direnç taşıyan E. gallinarum ve E. casseliflavus türlerinden özellikle E. gallinarum’un immün yetmezlik sorunu olan hastalarda bakteriyemi, endokardit ve menenjit gibi ciddi infeksiyonlara neden olabildiği bildirilmiştir (17). Bu türlerin doğru tanımlanması yalnızca tedavi için değil, aynı zamanda hastane içi bulaşmanın önlenmesi açısından da önemlidir.
Son yıllarda yurtdışında ve ülkemizde yapılan bazı çalışmalarda VanA tipi dirençli E. faecium etkenli VRE infeksiyonlarının oranlarında artış bildirilmektedir (18-22). Benzer şekilde, çalışmamızda da 2015–2019 yılları arasında izole edilen 38 klinik Enterococcus izolatı arasında, konvansiyonel kültür testleriyle %66, mPCR analizleriyle %61 oranında VanA dirençli E. faecium saptanmış ve bu tür en yüksek oranda belirlenmiştir.
Tür ve direnç belirlemede konvansiyonel testler ile mPCR yöntemi arasında 38 örneğin 31 (%82)’inde tam uyum saptanmıştır. Elde edilen sonuçlar, çalışmada kullanılan optimize mPCR testinin başarılı, hızlı, pratik ve ekonomik bir yöntem olduğunu göstermektedir. Konvansiyonel ve otomatize kültür sistemleri, VRE izolatlarının yalnızca vankomisine dirençli veya duyarlı olduğunu belirleyebilirken, mPCR testi direnç geninin tipini (vanA, vanB ve vanC-1) de ortaya koyabilmektedir. Bu durum tedavi seçimini değiştirmese de olası gen transferinin ve VRE yayılımının izlenmesi açısından klinik ve epidemiyolojik açıdan önem taşımaktadır.
Çalışmamızda iki yöntem arasında %18 oranında tanısal farklılık gözlenmesi tür ve direnç tespitinde konvansiyonel testler ile birlikte moleküler tanının önemli ve gerekli olduğunu göstermektedir. Tür düzeyinde karşılaştırıldığında özellikle E. gallinarum tanımlamasında farklılıklar saptandı. Kültür yöntemiyle E. faecium olarak tanımlanan bir izolat, E. casseliflavus olarak tanımlanan 2 izolat ve tanımlanamayan 3 izolat, mPCR yöntemiyle E. gallinarum olarak belirlendi.
Fang ve arkadaşları (23) tarafından yapılan bir çalışmada, Bruker MS, VITEK MS, VITEK 2 sistemleri ve mPCR yöntemi karşılaştırılmış; VITEK 2’nin 132 Enterococcus izolatın %91.7’sini tür düzeyinde doğru tanımlayabildiği bildirilmiştir. Ancak, 21 E. gallinarum izolatından 17’si doğru tanımlanmış, 9 izolat E. casseliflavus ile E. gallinarum arasında ayırt edilememiştir. VITEK 2 sisteminin E. faecalis ve E. faecium dışındaki Enterococcus türlerini tanımlamada daha az verimli olduğu bildirilmiştir (23). Başka bir çalışmada, VITEK 2 ve MALDI-TOF MS sistemleri ile 16S rRNA gen dizi analizi yöntemi karşılaştırılmış; E. gallinarum izolatlarının VITEK 2 sistemi tarafından doğru tanımlandığı, ancak, sistemin genel olarak daha düşük ayırt ediciliğe sahip olduğu ve 39 izolatın 10’unun yanlış tanımladığı bildirilmiştir. Örneğin, 6 Enterococcus durans izolatından 2’si E. gallinarum olarak yanlış tanımlanmıştır. Bu bulgular sonucunda çalışmada, otomatize tanımlama sistemlerinin benzer biyokimyasal özelliklere sahip türlerde sınırlı ayırt ediciliğe sahip olduğu ve veritabanlarının güncellenmesi gerektiği ifade edilmiştir (24).
Çalışmamızda, konvansiyonel kültür yöntemleriyle vankomisin dirençli olarak saptanan 3 izolatta mPCR yöntemiyle direnç geni saptanamadı. Benzer şekilde, literatürde de konvansiyonel ve moleküler yöntemlerin değerlendirildiği çalışmalarda direnç saptanamayan veya hatalı saptanan izolatlar bildirilmiştir. Bilen ve arkadaşları (22) tarafından yapılan bir çalışmada, otomatize identifikasyon sistemi ile tüm Enterococcus suşlarının vankomisin ve teikoplanine dirençli bulunduğu; ancak gradyan test yöntemine göre üç izolatın her iki antibiyotiğe de duyarlı olduğu, bu suşlarda vanA, vanB ve vanC genlerinin hiçbirinin saptanmadığı bildirilmiştir (25). Ayrıca bu durum, kullanılan moleküler testin kapsayıcılığına (örneğin vanC2-C3 gen bölgelerini hedeflemesi) bağlı olarak da değişebilir. Farklı PCR yöntemlerinin gen tespit kapasitelerindeki değişkenlik nedeniyle, direnç geni saptanamamış olabilir.
Bu bulgular, Enterococcus türlerine özgü genlerin ve antibiyotik direncinden sorumlu genlerin klinik laboratuvarlarda moleküler yöntemlerle saptanmasının yararlı olduğunu göstermektedir. Ancak yöntemin rutin kullanıma geçebilmesi için kitin maliyet etkinliği açısından geliştirilmesi ve kapsamlı validasyon çalışmaları gerekmektedir.
Yapılan çalışmalar, VRE’lerde yüksek düzeyde glikopeptid direncine neden olan genlerin, genellikle yüksek kopya sayısına ve yüksek stabiliteye sahip plazmidler ile bu plazmidlere entegre olmuş transpozonlar üzerinde bulunduğunu göstermektedir. Ayrıca bu direnç genleri, plazmid veya kromozomlara entegre olabilen konjugatif transpozonlar aracılığıyla da kazanılabilir. Bu transpozonlar, içerdikleri insersiyon dizileri aracılığıyla farklı konak DNA’lara entegre olma yeteneğine sahiptir (26). Literatürde, insan klinik örneklerinden hem vanC-1 hem vanA direnç genlerini birlikte taşıyan E. gallinarum (27-29) ve VanA dirençli E. faecalis (30) izolatlarının bildirildiği az sayıda çalışma bulunmaktadır. Çalışmamızda da benzer şekilde, özel bir direnç profili kapsamında sayılan VanC-1 ve vanA direnç birlikteliğine sahip bir E. gallinarum izolatı ve ayrıca vanB direnci yerine vanA direnci taşıyan bir E. faecalis izolatı saptandı. Bu bulgu son derece önemlidir; çünkü normalde VanC-1 fenotipi ile düşük düzeyde intrensek direnç göstermesi beklenen E. gallinarum türünde vanA tipi direnç geni bulunması ve orta düzey dirençten sorumlu vanB tipi direnç geni yerine vanA direnci taşıyan E. faecalis izolatı bulunması vanA direnç geninin ülkemizde de enterokok türleri arasında aktarılabildiğinin kanıtı niteliğindedir. Bu tür izolatların ayrıntılı olarak incelenmesi ve bu türlerin yayılımını önlemek amacıyla hastanelerde düzenli tarama programlarının sürdürülmesi gereklidir.
Sonuç olarak; bu çalışma, hastane infeksiyon etkeni klinik Enterococcus izolatları arasında en sık görülen E. faecium, E. faecalis ve E. gallinarum türlerinin ve bunların taşıdığı vanA, vanB ve vanC-1 direnç genlerinin, optimize edilmiş bir mPCR protokolü ile kısa sürede ve yüksek duyarlılıkla saptanabileceğini göstermiştir. Bu yöntem, VRE tarama ve sürveyans çalışmalarında etkin biçimde kullanılabilir.
Hasta Onamı
Veriler retrospektif olarak değerlendirildiği için hasta onamı alınmamıştır.
Etik Kurul Kararı
Bu çalışma içi için Ahi Evran Üniversitesi Girişimsel Olmayan Klinik Araştırmalar Etik Kurulu’ndan 20 Ekim 2020 tarih ve 2020-15/106 karar numarasıyla onay alınmıştır.
Danışman Değerlendirmesi
Bağımsız dış danışman
Yazar Katkıları
Fikir/Kavram – F.F.A.; Tasarım – F.F.A.; Denetleme – F.F.A., F.M.S.; Kaynak ve Fon Sağlama – F.F.A., F.M.S.; Malzemeler/Hastalar – F.M.S.; Veri Toplama ve/veya İşleme – F.F.A., F.M.S.; Analiz ve/veya Yorum – F.F.A.; Literatür Taraması – F.F.A., F.M.S.; Makale Yazımı – F.F.A., F.M.S.; Eleştirel İnceleme – F.F.A.
Çıkar Çatışması
Yazarlar herhangi bir çıkar çatışması bildirmemiştir.
Finansal Destek
Bu araştırma Kırşehir Ahi Evran Üniversitesi BAP Birimi tarafından TIP.A4.21.012 proje numarası ile desteklenmiştir.
Bilimsel Etkinlik
Bu çalışma, 7–9 Mart 2024 tarihlerinde Burdur’da düzenlenen 7. Uluslararası Sağlık Bilimleri ve Yaşam Kongresi (IHSLC 2024)’nde bildiri olarak sunulmuştur.
Referanslar
- de Kraker ME, Jarlier V, Monen JC, Heuer OE, van de Sande N, Grundmann H. The changing epidemiology of bacteraemias in Europe: trends from the European Antimicrobial Resistance Surveillance System. Clin Microbiol Infect. 2013;19(9):860-8. [CrossRef]
- Ranotkar S, Kumar P, Zutshi S, et al. Vancomycin-resistant enterococci: Troublemaker of the 21st century. J Glob Antimicrob Resist. 2014;2(4):205-12. [CrossRef]
- Faron ML, Ledeboer NA, Buchan BW. Resistance mechanisms, epidemiology, and approaches to screening for vancomycin-resistant Enterococcus in the health care setting. J Clin Microbiol. 2016;54(10):2436-47. [CrossRef]
- De Angelis G, Cataldo MA, De Waure C, et al. Infection control and prevention measures to reduce the spread of vancomycin-resistant enterococci in hospitalized patients: a systematic review and meta-analysis. J Antimicrob Chemother. 2014;69(5):1185-92. [CrossRef]
- Cattoir V, Leclercq R. Twenty-five years of shared life with vancomycin-resistant enterococci: is it time to divorce? J Antimicrob Chemother. 2013;68(4):731-42. [CrossRef]
- Werner G, Coque TM, Hammerum AM, et al. Emergence and spread of vancomycin resistance among enterococci in Europe. Euro Surveill. 2008;13(47):19046.
- Dutka-Malen S, Evers S, Courvalin P. Detection of glycopeptide resistance genotypes and identification to the species level of clinically relevant enterococci by PCR. J Clin Microbiol. 1995;33(5):1434. Erratum for: J Clin Microbiol. 1995;33(1):24-7. [CrossRef]
- Depardieu F, Perichon B, Courvalin P. Detection of the van alphabet and identification of enterococci and staphylococci at the species level by multiplex PCR. J Clin Microbiol. 2004;42(12):5857-60. [CrossRef]
- Teixeira LM, Carvalho GS, Facklam RR. Enterococcus. In: Murray PR, Baron EJ, Jorgensen JH, Landry ML, Pfaller MA, editors. Translated by Ö Akan. Başustaoğlu A, translation editor. Klinik Mikrobiyoloji (Manual of Clinical Microbiology). Ankara: Atlas Kitapçılık; 2009. p. 430-42.
- Gozalan A, Coskun-Ari FF, Ozdem B, et al. Molecular characterization of vancomycin-resistant Enterococcus faecium strains isolated from carriage and clinical samples in a tertiary hospital, Turkey. J Med Microbiol. 2015l;64(7):759-66. [CrossRef]
- Arı FF, Çopur Çiçek A, Çizmeci Z, Güldemir D, Balcı PÖ, Durmaz R. Vankomisine dirençli enterokoklarda tür ve direnç genlerinin (vanA, vanB ve vanC-1) multipleks PCR yöntemi ile saptanması. In: 7. Ulusal Moleküler ve Tanısal Mikrobiyoloji Kongresi Bildiri Özet Kitabı; 2012; Ankara, Türkiye. p. 255-6.
- European Committee on Antimicrobial Susceptibility Testing (EUCAST). Breakpoint tables for interpretation of MICs and zone diameters. Version 10.0, 2020 [Internet]. Växjö: European Committee on Antimicrobial Susceptibility Testing EUCAST. [erişim 30 Aralık 2023]. https://www.eucast.org
- Queipo-Ortuño MI, De Dios Colmenero J, Macias M, Bravo MJ, Morata P. Preparation of bacterial DNA template by boiling and effect of immunoglobulin G as an inhibitor in real-time PCR for serum samples from patients with brucellosis. Clin Vaccine Immunol. 2008;15(2):293-6. [CrossRef]
- Conly J, Shafran S. Emerging epidemiology of vancomycin-resistant enterococci in Canada. Can J Infect Dis. 1997;8:182-5.
- Arthur M, Molinas C, Depardieu F, Courvalin P. Characterization of Tn1546, a Tn3-related transposon conferring glycopeptide resistance by synthesis of depsipeptide peptidoglycan precursors in Enterococcus faecium BM4147. J Bacteriol. 1993;175(1):117-27. [CrossRef]
- Toye B, Shymanski J, Bobrowska M, Woods W, Ramotar K. Clinical and epidemiological significance of enterococci intrinsically resistant to vancomycin (possessing the vanC genotype). J Clin Microbiol. 1997;35(12):3166-70. Erratum in: J Clin Microbiol 1998;36(5):1469. [CrossRef]
- Centers for Disease Control and Prevention (US). Antibiotic resistance threats in the United States, 2013 [Internet]. Atlanta (GA): Centers for Disease Control and Prevention; 2013 [erişim 30 Aralık 2023]. https://www.cdc.gov/antimicrobial-resistance/media/pdfs/ar-threats-2013-508.pdf
- Aslan S, Öztürk C, Delialioğlu N, Emekdaş G. [Investigations on vancomycin and high level aminoglycoside resistance of enterococci isolates from clinical specimen]. Mersin Ünv Sağ Bil Derg. 2012;5(2):14-8. Turkish.
- Çelik C, Uysal EB, Gözel MG, Bakıcı MZ, Elaldı N. [Antimicrobial resistance patterns of Enterococcus faecalis and Enterococcus faecium bacteria isolated from bloodstream infections]. Flora. 2013;18(2):83-9. Turkish.
- Arbabi L, Boustanshenas M, Rahbar M, et al. Antibiotic susceptibility pattern and virulence genes in Enterococcus spp. isolated from clinical samples of Milad Hospital of Tehran, Iran. Arch Clin Infect Dis. 2016;11(3):e36260. [CrossRef]
- Sun HL, Liu C, Zhang JJ, Zhou YM, Xu YC. Molecular characterization of vancomycin-resistant enterococci isolated from a hospital in Beijing, China. J Microbiol Immunol Infect. 2019;52(3):433-42. [CrossRef]
- Bilen S, Parlak M, Yakupoğulları Y, et al. [Phenotypic and genotypic evaluation of vancomycin-resistant Enterococcus (VRE) strains isolated from various clinical specimens]. Turk Mikrobiyol Cemiy Derg. 2021;51(2):143-9. Turkish. [CrossRef]
- Fang H, Ohlsson AK, Ullberg M, Ozenci V. Evaluation of species-specific PCR, Bruker MS, VITEK MS and the VITEK 2 system for the identification of clinical Enterococcus isolates. Eur J Clin Microbiol Infect Dis. 2012;31(11):3073-7. [CrossRef]
- Kim SH, Chon JW, Jeong HW, et al. Identification and phylogenetic analysis of Enterococcus isolates using MALDI-TOF MS and VITEK 2. AMB Express. 2023;13(1):21. [CrossRef]
- Dombrádi Z, Bihari Z, Horváth KI, Szabó J. Comparison of the VITEK 2 system with the E-test for the determination of glycopeptide susceptibility of vanA and vanC positive enterococci. Acta Microbiol Immunol Hung. 2010;57(3):157-63. [CrossRef]
- Dunny GM, Leonard BA, Hedberg PJ. Pheromone-inducible conjugation in Enterococcus faecalis: interbacterial and host-parasite chemical communication. J Bacteriol. 1995;177(4):871-6. [CrossRef]
- Corso A, Faccone D, Gagetti P, et al. First report of VanA Enterococcus gallinarum dissemination within an intensive care unit in Argentina. Int J Antimicrob Agents. 2005;25(1):51-6. [CrossRef]
- Neves FP, Ribeiro RL, Duarte RS, Teixeira LM, Merquior VL. Emergence of the vanA genotype among Enterococcus gallinarum isolates colonising the intestinal tract of patients in a university hospital in Rio de Janeiro, Brazil. Int J Antimicrob Agents. 2009;33(3):211-5. [CrossRef]
- Togneri A, Lopardo H, Corso A. [Bacteremia caused by Enterococcus gallinarum with a high level of glycopeptide resistance: 1st documented cases in Argentina]. Rev Argent Microbiol. 2003;35(2):96-9. Spanish.
- Cereda R, Sader H, Sejas L, et al. Enterococcus faecalis resistant to vancomycin and teicoplanin (VanA phenotype) isolated from a bone marrow transplanted patient in Brazil. Braz J Infect Dis. 2001;5(1):40-6. [CrossRef]